Vol. XXXII Issue 2
Article 3
DOI: 10.35407/bag.2021.32.02.03
RESEARCH
Herramientas complementarias utilizadas en el programa de
mejoramiento de arveja (Pisum sativum l.) de la Universidad Nacional de Rosario
Complementary tools
utilized in the pea (Pisum sativum l.) breeding program at Universidad Nacional
de Rosario
Guindón
M.F.1
Cazzola
F.1
Bermejo
C.J.1
Espósito
M.A.1,3
Gatti I.2
Cointry E.L.1 *
1 Instituto de
Investigaciones en Ciencias Agrarias de Rosario, Consejo Nacional de Investigaciones
Científicas y Técnicas (IICAR-CONICET), Facultad de Ciencias Agrarias,
Universidad Nacional de Rosario (UNR), Zavalla, Argentina
2 CIUNR, Consejo de
Investigadores de la Universidad Nacional de Rosario, Rosario, Argentina;
3 Instituto Nacional
de Tecnología Agropecuaria, EEA INTA Oliveros, Ruta Nacional 11 km 353, Oliveros,
Santa Fe.
* Corresponding author: Cointry, E. L. ecointry@unr.edu.ar
ORCID 0000-0001-5906-7291
General Editor: Elsa Camadro
RESUMEN
El
mejoramiento convencional puede ser complementado mediante diferentes
estrategias que incrementen la eficiencia de las metodologías y la tasa actual
de aumento de los rendimientos a fin de satisfacer la demanda. El uso de
marcadores moleculares con el objetivo de desarrollar mapas de ligamiento de la
especie, el uso de Blup (Best Linear Unbiased Prediction) para una selección
eficiente de progenitores a hibridar, el uso del cultivo in vitro para
incrementar artificialmente el número de plantas F1 o el uso de
fenotipificación digital para una eficiente caracterización digital que puede
realizarse durante la regeneración periódica y rutinaria de accesiones en
colecciones de germoplasma.
Palabras clave: Marcadores
moleculares, Blup,
Cultivo in vitro, Fenotipificación
digital
ABSTRACT
Conventional breeding can be
complemented by different strategies that increase the efficiency of the
methodologies and the current rate of increase in yields in order to meet
demand. The use of molecular markers with the aim of developing linkage maps of
the species, the use of Blup (Best Linear Unbiased Prediction) for an efficient
selection of progenitors to hybridize, the use of in vitro culture to
artificially increase the number of F1 plants or the use of digital phenotyping
for efficient digital characterization that can be performed during the
periodic and routine regeneration of accessions in germplasm collections.
Key words: Molecular markers, Blup, In vitro
culture, Digital phenotyping
Received: 10/14/2020
Accepted: 10/29/2020
INTRODUCCIÓN
La
arveja (Pisum sativum L.) es una leguminosa autógama de estación fría y anual
que se origina en áreas del Medio Oriente, el este del Cáucaso, Irán y
Afganistán, y el oeste de la cuenca mediterránea (Smýkal et al., 2011). Su genoma está
organizado en siete pares de cromosomas (2n=2x=14) y el tamaño del genoma
haploide se estima en 4,45 Gb (Smýkal et al., 2012). Fueron y son hasta el día
de hoy una fuente importante de alimento para animales y humanos. La especie es
rica en proteínas, almidón de digestión lenta, azúcares solubles, fibra,
minerales y vitaminas (Dahl et al., 2012).
Teniendo
en cuenta que el aumento de la población mundial requerirá una mayor producción
de cultivos es necesario entonces, incrementar la tasa actual de aumento de los
rendimientos para satisfacer esta demanda (Tester y Langridge,
2010).
Para ello pueden recurrirse a diferentes estrategias.
Utilización de marcadores moleculares
En
este contexto, el desarrollo de mapas de ligamiento se constituye en una herramienta
útil para maximizar la probabilidad de éxito, ya que son herramientas poderosas
para la investigación genética y el mejoramiento, y son el primer paso en: a)
el análisis de rasgos cualitativos y cuantitativos; b) la introgresión de genes
deseables y loci de rasgos cuantitativos (QTL); y c) clonación posicional o
basada en mapas de genes responsables de rasgos económicamente importantes (Semagn
et al., 2006).
Diferentes tipos de marcadores se han utilizado para desarrollar mapas de
ligamiento de densidad moderada en arveja. Loridon et al. (2005), Sun
et al. (2014)
y Yang
et al. (2015)
utilizaron SSR (Simple Sequence Repeats); Deulvot et al. (2010) utilizaron SNP
(Single Nucleotide Polymorphisms); Mishra et al. (2009) emplearon ISSR
(Inter Simple Sequence Repeats) y Barilli et al. (2010) construyeron sus
mapas usando STS (Sequence Tagged Sites). Nosotros propusimos el uso de
Sequence Related Amplified Polymorphism (SRAP) (Li y Quiros, 2001) para generar una
serie de marcadores distribuidos en todos los cromosomas de arveja. Desde su
desarrollo, estos marcadores se han empleado en una amplia gama de especies
para la estimación de la diversidad genética (Bermejo et al., 2014; Zheng
et al., 2017;
Kumar y Agrawal, 2017), en la construcción de mapas (Martin
et al., 2013;
Padmakar
et al., 2015)
y en análisis QTL (Liu et al., 2016; Martin et al.,
2016).
Para
su desarrollo se generó una población de mapeo F2 derivada de un cruzamiento
inicial entre dos cultivares DDR11 (The Indian Council of Agriculture Research)
y Zav25 (material local derivado de nuestro programa) que son contrastantes
para la mayoría de las características relacionadas con el rendimiento, como el
número de vainas y semillas por parcela. Se evaluaron un total de 25
combinaciones de cebadores SRAP en plantas F2 y en ambas líneas parentales,
generando 208 bandas/marcadores polimórficos. Este primer mapa de ligamiento (Guindón
et al., 2016)
constó de 112 marcadores genéticos distribuidos en 7 grupos de ligamiento (LG),
que cubren un total de 528,8 cM. La longitud de los LG varió de 47,6 a 144,3 cM
(media 75,54 cM), con 9 a 34 marcadores.
Posteriormente
y en un trabajo en conjunto con el Dr. Thomas Warkentin del Crop Development
Centre, Department of Plant Sciences, de la Universidad de Saskatchewan,
(Canadá), se profundizó en el desarrollo de un mapa de ligamiento genético
usando SRAP, SSR y SNP para identificar QTL que controlan los caracteres
relacionados con el rendimiento. Una población F2 y sus familias F2:3 derivadas
de un cruce inicial entre cvs. Explorer (desarrollada por Svalof Weibull en
Suecia), variedad a semi-áfila, con color verde de cotiledón y vainas y granos
de tamaño intermedio y DDR14 (desarrollada por Indian Council of Agriculture
Research), foliosa, con cotiledón color amarillo, vainas y granos de gran
tamaño, fueron evaluadas con SRAP, SSR y técnicas GBS, que demostraron ser
eficientes, generando un conjunto de 872 marcadores polimórficos para mapeo de
ligamiento. El mapa resultante constó de 128 genes marcadores distribuidos en 9
grupos de ligamiento (LG) (Figura 1 a y b), que cubrieron 655,5 cM. La
longitud de los LG osciló entre 49,1 y 114,8 cM, con 8 a 26 marcadores. La
detección de QTL fue realizado utilizando el método mapeo de intervalo
compuesto (CIM). Se detectaron un total de 45 QTL a través del generaciones y
ambientes evaluados. Todos ellos fueron QTL importantes que explicaron más del
10% de la variación fenotípica (Guindón et al., 2019b).

Figura 1. Mapa de ligamiento, con nombres de los
marcadores a la derecha de cada LG (grupo de ligamiento) y distancias del mapa
(en cM) a la izquierda. Marcadores con niveles significativos de distorsión por
segregación se indican con asteriscos. (Figura tomada de Guindón
et al. 2016
CBAB)
Estos
estudios nos permiten aplicar estos conocimientos en el programa de mejora
asistida por marcadores moleculares.
Selección eficiente de progenitores a hibridar: Uso de
BLUP
Diferentes
criterios han sido empleados para la selección de variedades a hibridar para
generar una población F2 susceptible de ser seleccionada. Un primer criterio
está basado en la explotación de la heterosis para diferentes caracteres
agronómicos luego de la hibridación ya que existe una correlación positiva
entre heterosis y alta frecuencia de líneas recombinantes transgresivas (Rieseberg
et al., 1999;
Espósito
et al., 2014;
Joshi
et al., 2015,
Guindón
et al., 2018).
Un segundo criterio consiste en la selección de variedades basado en sus
valores fenotípicos, pero no es el más adecuado al estar estos valores
fenotípicos influenciados por el efecto de las desviaciones ambientales. Un
tercer criterio está basado en la utilización de valores genotípicos. Lorenzana
y Bernardo (2009)
evaluaron la precisión de diferentes métodos para la predicción de valores genotípicos
y concluyeron que el enfoque BLUP (Best Linear Unbiased Prediction) es el mejor
método para predecir los valores genotípicos en poblaciones biparentales ya que
se corrigen mejor las variaciones extrañas, se pueden analizar datos
desbalanceados y se puede incorporar la información del pedigrí (Piepho
et al., 2008).
Guindón et al. (2018) aplicaron BLUP para la predicción de valores genotípicos
utilizando datos morfológicos de diferentes años correspondientes a las
variedades progenitoras, la generación F1 y las poblaciones F3 ya que la
población F2 al no poderse replicar fue imposible establecer el modelo lineal
para la obtención de los valores BLUP. Este análisis permitió la corrección por
efectos ambientales. Estas estimaciones se utilizaron para el análisis genético
de diferentes caracteres. Se observó heterosis para número de vainas (27,1%) y
número de semillas (23,3%), caracteres que tienen un efecto directo sobre el
rendimiento. Un análisis de componentes principales (PCA) se realizó utilizando
datos de BLUP para obtener una representación gráfica de la estructura de
relaciones de las 110 familias F3 (Figura 2). Se calcularon las
distancias de Manhattan y la matriz de distancias se sometió a un análisis de
agrupamiento utilizando el promedio ponderado.
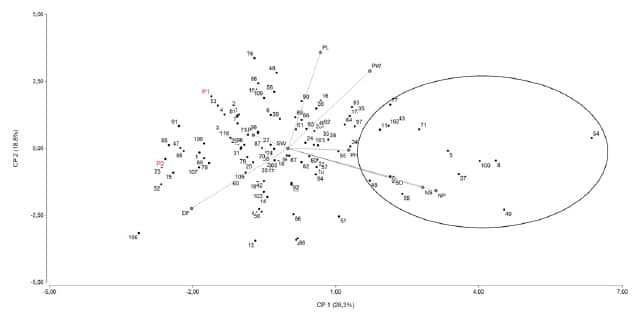
Figura 2. Relaciones entre las 110 familias F3
derivadas de Explorer (P2) y DDR14 (P1) en base al análisis de componentes
principales. Las familias dentro del círculo son transgresivas para número de
semillas y vainas. La posición de los padres es indicada en rojo.
Mediante
el uso de BLUP y de un análisis de componente principales, fue posible elegir
familias con buen desempeño lo que muestra la importancia de estas metodologías
para los programas de mejora de arveja.
Uso del cultivo in vitro
En
los programas de mejoramiento se requiere una gran cantidad de individuos F2
para realizar el proceso de selección correctamente, pero a menudo hay pocas
plantas disponibles ya que ciertos cruzamientos producen pocas semillas F1,
probablemente debido a diferencias en la estructura de la flor que dificultan
la castración manual. Para obtener más semillas F2, es necesario entonces
proceder a incrementar artificialmente el número de plantas F1. Se han
reportado estudios de regeneración in vitro en arveja, utilizando diferentes
explantos, como nudos cotiledonales (Rajput y Singh, 2010), cotiledones (Pniewsky
et al., 2003),
folíolos inmaduros (Fujioka et al., 2000), embriones
cigóticos (Sánchez y Mosquera, 2006) y semillas maduras (Zhihui
et al., 2009).
Espósito
et al. (2012)
desarrollaron un protocolo rápido, eficiente y reproducible para la regeneración
de brotes in vitro y el enraizamiento de dichos vástagos para la obtención de
múltiples genotipos F1 usando semillas maduras (Figura 3).

Figura 3. Múltiples vástagos desarrollados de la
semilla original. de la enfermedad (Porob et al., 2017).
Las
plántulas regeneradas se trasplantaron al suelo en macetas, encontrándose que
el 60% de los vástagos regenerados enraizaron en un período de seis semanas.
Generalmente, las tasas de mortalidad se debieron a tallos y raíces débiles que
no pudieron adquirir los nutrientes del suelo cuando son transferidas del medio
agarizado. Las plántulas que desarrollaron normalmente se trasplantaron a
macetas y en invernadero cultivándose hasta la madurez. Este procedimiento se
puede utilizar en programas de mejoramiento lo que permitirá trabajar con más
plantas siempre que los cruces tengan poca producción de semillas.
Uso de fenotipificación digital
La
fenotipificación de plantas vincula la genómica con la ecofisiología y la agronomía.
Por lo general, se realiza mediante tecnología no destructiva, automatizada y
basada en imágenes, y genera información para la eficiente caracterización
digital y que se puede realizar durante la regeneración periódica y rutinaria
de accesiones en colecciones de germoplasma. Gatti et al. (2017) estudiaron 92
accesiones del género Pisum de diferentes especies y subespecies durante dos
ciclos de cultivo midiendo caracteres asociados al tamaño y color de semillas y
vainas utilizando imágenes digitales. Estas características obtenidas por
fenotipificación digital, demostraron ser marcadores adecuados para la
evaluación de la diversidad genética y útiles en el análisis evolutivo,
permitiendo la discriminación de las principales especies silvestres y cultivadas
del género Pisum. También se la ha utilizado para fenotipificar varios
parámetros del estado fitosanitario, como contenido de clorofila (Dutta
Gupta et al., 2013),
contenido de nitrógeno (Vollmann et al., 2011) y estado de la
enfermedad (Porob et al., 2017).
Durante
el almacenamiento de la arveja pueden ocurrir pérdidas significativas en el
color, debido a la pérdida de clorofila o al blanqueado de las semillas y este
deterioro influye en la comercialización y en la decisión de compra por parte
de los consumidores. La magnitud de esta pérdida de color puede ser estudiada a
partir de la técnica de envejecimiento acelerado, que implica someter a las
semillas a condiciones severas de temperatura y humedad relativa y usar el
fenotipificación digital es para medir dicho deterioro. Guindón
et al. (2019
a), demostraron la existencia de un comportamiento diferencial de las
variedades en el mantenimiento del color verde de las semillas mediante esta metodología,
pudiendo así también ser aplicada en procesos de selección durante la obtención
de nuevas variedades.
BIBLIOGRAFÍA
Barilli E., Satovic Z., Rubiales D.,
Torres A.M. (2010) Mapping of quantitative trait loci controlling partial
resistance against rust incited by Uromyces pisi (Pers.) Wint. in a Pisum
fulvum L. intraspecific cross. Euphytica 175: 151-159.
Bermejo C., Gatti I., Caballero N.,
Cravero V., Martin E., Cointry E. (2014) Study of diversity in a set of lentil
RILs using morphological and molecular markers. Aust. J. Crop Sci. 8: 689.
Dahl W., Foster L., Tyler R. (2012)
Review of the health benefits of peas (Pisum sativum L.). British J. Nutrition 108:
3-10.
Deulvot C., Charrel H., Marty A.,
Jacquin F., Donnadieu C., Lejeune Hénaut I., Burstin J., Aubert G. (2010) Highlymultiplexed
SNP genotyping for genetic mapping and germplasm diversity studies in pea. BMC
Genomics 11: 468- 478.
Dutta Gupta S., Ibaraki Y., Pattanayak
A.K. (2013) Development of a digital image analysis method for real-time
estimation of chlorophyll content in micropropagated potato plants. Plant
Biotechnol. Rep. 7: 91-97.
Espósito M.A., Almirón P., Gatti I.,
Cravero V., López Anido F., Cointry E.L. (2012) A rapid method to increase F1
plants number in breeding pea programs (Pisum sativum L.). Genet. Mol. Res. 1: 2729-2732.
Espósito M.A., Bermejo C., Gatti I.,
Guindón M.F., Cravero V., Cointry E.L. (2014) Prediction of heterotic crosses
for yield in Pisum sativum L. Sci. Hortic. 177: 53-62.
Fujioka T., Fujita M., Iwamoto K.
(2000) Plant regeneration of Japanese pea cultivars by in vitro culture of
immature leaflets. J. Jpn. Soc. Hortic. Sci. 69: 656-658.
Gatti I., Guindón F., Bermejo C.,
Cointry E.L. (2017) Analysis of variability and phylogeny in pisum (Pisum spp.)
using digital phenotyping and morphological traits. AJCS 11 (12): 1599-1605.
Guindón M.F., Agüero M.G., Cointry
E. Evaluación
del cambio de color en arveja (Pisum sativum L.) por efecto del envejecimiento
acelerado. Comunicaciones Libres XXI Congreso y XXXIX Reunión Anual de la
Sociedad de Biología de Rosario, 26- 27 noviembre 2019 (a), Rosario, Santa Fe,
p. 57.
Guindón M.F., Martin E., Cravero V.,
Cointry E. (2018) Transgressive segregation, heterosis and heritability for
yield related traits in a segregating population of Pisum sativum L. Exp. Agric.
55: 610-620.
Guindón F., Martín E., Cravero V.,
Gali Krishna K., Warkentin T.D., Cointry E.L. (2019b) Linkage map development
by G BS, SSR and SRAP techniques and yield-related QTLs in pea. Mol. Breed. 39:
54-70.
Guindón M.F., Martin E., Zayas A.,
Cointry E., Cravero V. (2016) Evaluation of SRAP markers for mapping of Pisum
sativum L. Crop Breed. Appl. Biotech. 16: 182-188.
Joshi D.J., Ravindrababu Y., Patel
A.M., Chauhan S.S. (2015) Heterosis studies for grain yield and its
contributing traits in field pea (Pisum sativum (L.) var. arvense). Asian J.
Bioscience 10: 158-161.
Kumar J., Agrawal V. (2017) Analysis
of genetic diversity and population genetic structure in Simarouba glauca DC.
(an important bio-energy crop) employing ISSR and SRAP markers. Ind. Crop Prod.
100: 98-207.
Li G., Quiros C.F. (2001) Sequence-related
amplified polymorphism (SRAP), a new marker system based on a simple PCR
reaction: its application to mapping and gene tagging in Brassica. Theor. Appl.
Genet. 103: 455-461.
Liu Z., Bao D., Liu D. (2016) Construction
of a genetic linkage map and QTL analysis of fruit-related traits in an F1 red
fuji xhongrou apple hybrid. Open Life Sci. 11: 487-497.
Lorenzana R.E., Bernardo R. (2009)
Accuracy of genotypic value predictions for marker-based selection in
biparental plant populations. Theor. Appl.
Genet . 120: 151-161.
Loridon K., McPhee K., Morin J., Dubreuil
P., Pilet Nayel M.L., Aubert G., Rameau C., Baranger A., Coyne C., Lejeune
Henaut I., Burstin J. (2005) Microsatellite marker polymorphism and mapping in
pea (Pisum sativum L.). Theor. Appl. Genet. 111:
1022-1031.
Martin E.A., Cravero V.P., Lopez
Anido F., Cointry E.L. (2016) QTLs detection and mapping for yield-related
traits in globe artichoke. Sci. Hortic. 202: 156-
164.
Martin E., Cravero V., Portis E.,
Scaglione D., Acquaviva E., Cointry E. (2013) New genetic maps for globe
artichoke and wild cardoon and their alignment with an SSR-based consensus map. Mol. Breed. 32: 177-187.
Mishra R.K., Kumar A., Chaudhary S.,
Kumar S. (2009) Mapping of the multifoliate pinna (mfp) leaf-blade morphology
mutation in grain pea Pisum sativum. J. Genet. 88: 227-232.
Padmakar B., Kanupriya C., Latha P.M.,
Prashant K.S., Dinesh M.R., Sailaja D., Aswath C. (2015) Development of SRAP
and SSR marker-based genetic linkage maps of guava (Psidium guajava L.). Sci. Hortic. 192: 158-165.
Piepho H.P., Möhring J., Melchinger
A.E., Büchse A. (2008) BLUP for phenotypic selection in plant breeding and
variety testing. Euphytica 161: 209-228.
Pniewsky T., Wachowiak J., Kapusta
J., Legocki A. (2003) Organogenesis and long-term micropropagations polish pea
cultivars. Acta Soc. Bot. Pol. 72: 295-302.
Porob S., Naik G., Velingkar H., Amonkar
D., Patil R., Bhat P. (2017) Plant Health Monitoring using Digital Image
Processing. IJETED 7 (3): 147-151.
Rajput V., Singh N.P. (2010) Studies
on in vitro regeneration and direct organogenesis in pea (Pisum sativum L.). Indian
J Plant Physiol. 15: 246-249.
Rieseberg L., Archer M., Waynw R.
(1999) Transgressive segregation, adaptation and speciation. Heredity 83: 363-372.
Sanchez E.A., Mosquera T. (2006) Establishing
a methodology for inducing the regeneration of pea (Pisum sativum L.) explants,
‘Santa Isabel’ variety. Agron. Colomb. 24: 17- 27.
Semagn K., Bjørnstad A., Ndjiondjop
M. (2006) An overview of molecular marker methods for plants. Afr J. Biotech. 5
(25):2540-2568.
Smýkal P., Kenicer G., Flavell A.J.,
Corander J., Kosterin O., Redden R.J., Ford R., Coyne C.J., Maxted N., Ambrose M.J.,
Ellis N.T.H. (2011) Phylogeny, phylogeography and genetic diversity of the
Pisum genus. Plant Genet. Res.: Characterization and Utilization 9: 4-18.
Smýkal P., Aubert G., Burstin J.,
Coyne C., Ellis N., Flavell A., Ford R., Hýbl M., Macas J., Neumann P., McPhee K.,
Redden R., Rubiales D., Weller J., Warkentin T. (2012) Pea (Pisum sativum L.)
in the Genomic Era. Agronomy 2: 74-115.
Sun X., Yang T., Hao J., Zhang X.,
Ford R., Jiang J., Wang F., Guan J., Zong X. (2014) SSR genetic linkage map
construction of pea (Pisum sativum L.) based on Chinese native varieties. Crop
J. 2: 170-174.
Tester M., Langridge P. (2010) Breeding
technologies to increase crop production in a changing world. Science 327: 818-822.
Vollmann J., Walter H., Sato T., Schweiger
P. (2011) Digital image analysis and chlorophyll metering for phenotyping the
effects of nodulation in soybean. Comput. Electron Agric. 75: 190-195.
Yang T., Fang L., Zhang X., Hu J.,
Bao S., Hao J., Li L., He Y., Jiang J., Wang F., Tian S. (2015) High-throughput
development of SSR markers from pea (Pisum sativum L.) based on next generation
sequencing of a purified Chinese commercial variety. PLoS One 10: 1-14.
Zheng Y., Xu S., Liu J., Zhao Y.,
Liu J. (2017) Genetic diversity and population structure of Chinese natural
bermudagrass (Cynodon dactylon L. Pers.) germplasm based on SRAP markers. PLoS
One 12:
e0177508. https://doi.org/10.1371/journal.pone.0177508.
Zhihui S., Tzitzikas M., Raemakers
K., Zhengqiang M. (2009) Effect of TDZ on plant regeneration from mature seeds
in pea (Pisum sativum). In Vitro Cell Dev. Biol. 45: 776-782.



