Vol. XXXII Issue 2
Article 5
DOI: 10.35407/bag.2021.32.02.05
RESEARCH
Mejoramiento de la calidad del fruto por la incorporación
de genes de especies silvestres en el tomate (Solanum lycopersicum L.)
Fruit quality
improvement through the incorporation of wild species genes in the tomato
(Solanum lycopersicum L.)
Pereira
da Costa J.H.1,2
Cambiaso
V.1
Picardi
L.A.1,3
Pratta
G.R.1,2
Rodríguez
G.R.1,2
1 Cátedra de Genética,
Facultad de Ciencias Agrarias, Universidad Nacional de Rosario. Campo
Experimental Villarino, (S2125ZAA) Zavalla, Santa Fe, Argentina.
2 Instituto de
Investigaciones en Ciencias Agrarias de Rosario (IICAR-CONICET-UNR), Campo
Experimental Villarino, (S2125ZAA) Zavalla, Santa Fe, Argentina .
3 Consejo de
Investigaciones de la Universidad Nacional de Rosario (CIUNR) . Universidad Nacional de Rosario. Maipú 1065, (S2000CGK)
Rosario, Santa Fe, Argentina.
* Corresponding
author:
Gustavo R. Rodríguez. grodrig@unr.edu.ar ORCID 0000-0002-4171-4099
General Editor: Elsa Camadro
RESUMEN
En
el mejoramiento del tomate (Solanum lycopersicum L.) se ha logrado un
incremento significativo para el rendimiento y otras características
productivas en un período corto de tiempo. Como consecuencia se redujo
notablemente la diversidad genética. Si bien el germoplasma silvestre se ha
utilizado principalmente como fuente de genes de resistencia para enfermedades
y plagas, nuestro grupo inició en la década de 1990, un programa de
mejoramiento genético en tomate para mejorar la calidad del fruto con especial
énfasis en incrementar la vida poscosecha y también ampliar la variabilidad
genética con la incorporación de estos genes al gran cultivo. Hemos
desarrollado diferentes poblaciones a partir del cruzamiento interespecífico
entre el cultivar argentino Caimanta de S. lycopersicum y la accesión LA0722 de
S. pimpinellifolium L. Mediante la generación de cruzamientos entre estos
padres selectos y el posterior avance generacional de la selección se ha
tratado de dilucidar las bases genéticas que definen la calidad del fruto. Para
ello se integraron al programa de mejoramiento información obtenida de datos
genómicos, posgenómicos y bioinformáticos. Al mismo tiempo hemos desarrollado
cuatro nuevos cultivares con características de calidad de fruto superiores al
ser comparados con híbridos comerciales. Para conservar y estudiar la
diversidad del cultivo también estamos desarrollado una colección de
germoplasma que en la actualidad cuenta con 162 genotipos de tomate de
diferentes especies y orígenes. Además, se ha iniciado la transferencia directa
de plantines a huertas urbanas y periurbanas para favorecer el acceso a
semillas de estos cultivares desarrollados en instituciones públicas.
Palabras clave: Solanum
pimpinellifolium, Vida poscosecha de los
frutos, Marcadores moleculares, Banco de germoplasma,
Huertas agroecológicas
ABSTRACT
The genetic improvement of tomato
(Solanum lycopersicum L.) has achieved an increase for yield and other
agronomic traits in a short period of time. As a consequence, genetic diversity
has been notably reduced. Wild germplasm has been mostly used as a source of
resistance genes for diseases and pests. Our group started in the 1990’ a
breeding program in tomato for improving fruit quality, with special emphasis
on increasing fruit shelf life and broadening the genetic variability with the
incorporation of wild genes. We have developed different populations from the
interspecific cross between the Argentine cultivar Caimanta of S. lycopersicum
and the accession LA0722 of S. pimpinellifolium L. Through crosses between
these selected parents and the subsequent generational selection advance, we
attempted to elucidate the genetic bases that underlie tomato fruit quality. To
do that, we use state-of-the-art technology available in the field of genetics
and breeding programs, including genomic, post-genomic and bioinformatic data.
At the same time, we have developed four new cultivars with improved fruit
quality traits compared to commercial hybrids. To conserve and study the tomato
diversity, we have developed a germplasm collection that currently contains 162
tomato genotypes from different species and origins. In addition, we have
started a direct transfer of our cultivars to urban and peri-urban community
orchards to facilitate them the access to genotypes that were developed in
Argentine public institutions.
Key words: Solanum pimpinellifolium, Fruit shelf life, Molecular
markers, Germplasm bank, Agroecological orchards
Received: 07/16/2021
Revised version received: 08/26/2021
Accepted: 09/01/2021
SOBRE EL CULTIVO DEL TOMATE
Origen y domesticación
El
tomate cultivado (Solanum lycopersicum L.) pertenece a la familia de las
solanáceas que incluye a más de 3.000 especies, muchas con importancia
económica como la papa, berenjena, petunia, tabaco y pimiento, entre otras. Es
originario de América y las 12 especies silvestres relacionadas se encuentran
distribuidas desde el centro de Ecuador, pasando por Perú hasta el norte de
Chile como así también en las Islas Galápagos (Spooner et al., 2005). Estas especies
crecen en una gran variedad de hábitats desde el nivel del mar a lo largo de la
costa del Pacífico a más de 3.300 metros en los valles del lado occidental de
los Andes, desde climas áridos a climas lluviosos. La gran diversidad
encontrada en las especies silvestres se expresa tanto en características
morfológicas, como fisiológicas y sexuales, y las convierte en recursos
genéticos muy valiosos. La primera fase de domesticación habría ocurrido en
manos de los primeros agricultores en Ecuador o el norte de Perú y luego
selecciones adicionales habrían tenido lugar en México a partir de poblaciones
predomesticadas y nuevamente en Sudamérica luego de una reintroducción (Razifard
et al., 2020;
Blanca
et al., 2021).
Desde Centroamérica, el tomate fue llevado a Europa por los españoles y luego
se extendió por todo el mundo. Se considera que S. pimpinellifolium L. sería el
antecesor silvestre más cercano del tomate cultivado y que S. lycopersicum var.
cerasiforme es un intermediario evolutivo entre S. pimpinellifolium y el
cultivado. Desde una perspectiva evolutiva, la domesticación y la selección
artificial a manos del hombre indujo importantes cambios fisiológicos y redujo
notablemente su diversidad genética. Las consecuencias más importantes de este
proceso han sido la modificación del sistema reproductivo, el incremento del
tamaño del fruto y la reducción de la base genética (Warnock,
1991).
Historia del mejoramiento del cultivo
Los
primeros registros que describen los intentos de seleccionar los mejores tipos
varietales fueron realizados en Europa en la segunda mitad del siglo XIX.
Durante los primeros treinta años del siglo XX, se fundaron varias empresas de
mejoramiento y producción de semillas de tomate que se establecieron por todo
el mundo. El mejoramiento de las variedades adaptadas a la cosecha mecánica se
inició en 1943, pero la liberación al mercado recién ocurrió en la década de
1960 (Rasmussen, 1968). Esta especialización del mercado ha
llevado a la diferenciación de las variedades de tomate que hoy se producen (Sim
et al., 2009).
En la década de 1940 se liberaron al mercado los cultivares híbridos de tomate
que garantizaban uniformidad de plantas y frutos, resistencia y altos
rendimientos. Estudios recientes han demostrado la recuperación de la riqueza
alélica a partir de la década de 1960 con la introgresión de genes para
resistencia a enfermedades e insectos desde S. peruvianum L., S. pennellii
Correll, S. chilense Dunal Reiche y S. habrochaites Knaap & Spooner. En la
década de 1980 también se incorporaron genes alternativos para tamaño de fruto,
color y sabor (Sim et al., 2012; Schouten et al., 2019).
En
Argentina, recién en 1946 se comenzó un plan de mejora en tomate y según Gallardo
(2012),
es muy probable que las selecciones obtenidas de tomate platense (Cattáneo
et al., 2020)
y las realizadas en la EEA Tucumán, hayan sido las primeras resultantes de la
aplicación de un método científico. En la actualidad, el país posee un perfil
netamente importador de semillas de tomate. En tomate se comercializan dos
tipos de estructuras genéticas: híbridos F1 o líneas puras o variedades. El
desarrollo de nuevos genotipos ha quedado relegado a las universidades y las
estructuras de I+D públicas, tal es así que de los 351 genotipos registrados en
los últimos 10 años ante el Instituto Nacional de Semillas (INASE), sólo hubo 12
variedades desarrolladas en nuestro país. De estas, diez son variedades o
cultivares mejorados cuyo objetivo de producción es la industria y la
Institución que tiene los derechos de obtentor es el INTA y las dos restantes
son cultivares mejorados para destinar la producción al consumo en fresco cuyo
derecho de obtentor pertenece a la Facultad de Ciencias Agrarias de la
Universidad Nacional de Rosario. Las semillas importadas satisfacen los
requerimientos buscados por los productores locales pero, además del alto
costo, son cultivares que fueron desarrollados para otros ambientes y
condiciones de cultivo y, sobre todo, carecen de la calidad demandada por los
consumidores.
Producción mundial y nacional de tomate
La
producción mundial de tomates frescos ha sido liderada desde 1960 por China en
Asia, por Italia y España en Europa y por los Estados Unidos, México y Brasil
en el continente americano. La misma ha aumentado sostenidamente desde mediados
de los años 1990 hasta superar en 2019 los 180 millones de toneladas a nivel
mundial y los 6,5 millones de toneladas en Sudamérica. En nuestro país la
producción se ha mantenido estable desde 1996 a la fecha en valores cercanos a
1,1 millones de toneladas anuales, cosechadas en unas 17 mil ha productivas
(FAO STAT, 2021). Aproximadamente el 60-70% de la producción nacional se
destina a consumo en fresco mientras que el 30-40% restante, a la elaboración
de productos industrializados. Esta actividad está concentrada principalmente
en la región de Cuyo y algunos años resulta deficitaria requiriendo
generalmente de la importación de productos desde Chile e Italia. En cambio, el
mercado interno nacional se mantiene provisto de tomates para consumo en fresco
durante todo el año a pesar de la estacionalidad del cultivo. Esto se logra
gracias a la amplia distribución de las zonas productivas que van desde Salta y
Jujuy, al Sur de la provincia de Buenos Aires y a las diferentes estrategias de
manejo aplicadas (Ministerio de Agricultura, Ganadería y Pesca, 2020).
El
Cinturón Hortícola de Rosario (CHR) se destaca no sólo por su nivel de
producción, sino también por la comercialización, ya que concentra y abastece a
más de 2 millones de habitantes. Por convicción comunitaria o imposición
legislativa, la zona productiva lindante o integrada a la ciudad de Rosario,
así como también a otras ciudades del país en las que existen producciones
hortícolas se han convertido recientemente a la agroecología. Este sistema de
producción amerita la generación de nuevos conocimientos y desarrollos
tecnológicos.
PROGRAMA DE MEJORAMIENTO PARA CALIDAD DE FRUTO EN LA
UNIVERSIDAD NACIONAL DE ROSARIO
Este
programa se inició en la década de 1990 para mejorar la calidad del fruto en
tomate, con especial énfasis en incrementar la vida poscosecha y ampliar la
variabilidad genética con la incorporación de genes silvestres al gran cultivo.
La vida poscosecha (Vp), definida como los días que transcurren desde la
cosecha del fruto cuando se visualiza el cambio de color en superficie hasta
que el mismo es comercialmente inaceptable por presentar síntomas de deterioro
tales como arrugamiento o exceso de ablandamiento, es un carácter de
fundamental importancia cuando el fruto de tomate se destina al consumo en
fresco. Las estrategias de mejoramiento para extender este carácter se basan en
la utilización de parentales que portan mutaciones que alteran la madurez.
Mutantes tales como el nor (non ripening), rin (ripening inhibitor) y cnr
(colorless non-ripening) exhiben efectos pleiotrópicos indeseables sobre el
color, la textura y la jugosidad del fruto incluso en condición heterocigota (Vrebalov
et al., 2002;
Manning et al., 2006; Wang et al., 2019). Otra estrategia
que retrasa la maduración del fruto es el uso de la ingeniería genética que ha
buscado modificar las proteínas de la pared celular (Brummell
y Harpster, 2001;
Meli
et al., 2010;
Uluisik
et al., 2016;
Yang
et al., 2017)
o incluso editar genéticamente estos genes mutantes por la metodología
CRISPR/Cas9 (Yu et al., 2017). Aunque estas
estrategias han tenido éxito científico, carecen de la aceptación del
consumidor y no son aceptadas por leyes existentes en diferentes países (Ishii
y Araki, 2016;
Schmidt
et al., 2020).
Hemos
demostrado que los frutos de las formas silvestres de S. pimpinellifolium y S.
lycopersicum var. cerasiforme tienen mayor vida poscosecha que los cultivares
comerciales de tomate (aunque menor que los genotipos homocigotos para los
mutantes de madurez del fruto tales como los ya mencionados nor y rin). Además,
presentan para este carácter una acción génica de tipo aditivo lo que permite
una respuesta a la selección (Zorzoli et al., 1998; Pratta
et al., 2000;
Rodríguez
et al., 2006a;
Rodríguez et al., 2010; Pereira da Costa et al., 2013; Cambiaso
et al., 2019a).
En este contexto se han desarrollado diferentes poblaciones a partir del
cruzamiento interespecífico entre el cultivar argentino Caimanta de S.
lycopersicum y la accesión LA0722 de S. pimpinellifolium: poblaciones F2,
líneas endocriadas recombinantes (RIL, Recombinant Inbred Lines), Híbridos de
Segundo Ciclo (HSC) y líneas casi isogénicas (NIL, Near Isogenic Lines)
(Rodríguez et al., 2006b; Cabodevila et al., 2017; Di
Giacomo et al., 2020).
Con
la generación de cruzamientos entre estos padres y el avance de la selección
generacional se han realizado distintas experiencias para dilucidar las bases
genéticas involucradas en los caracteres que definen la calidad del fruto
contando con la generación y utilización de datos genómicos, posgenómicos y
bioinformáticos.
MÉTODOS DE MEJORAMIENTO UTILIZADOS
Obtención de Líneas Endocriadas Recombinantes (RIL) por
el método genealógico.
La
población base fue la F2 del cruzamiento interespecífico entre el cultivar Caimanta
de S. lycoperscicum y la línea LA0722 de S. pimpinellifolium. El cultivar fue
provisto por la Estación Experimental Agropecuaria de INTA Cerrillos, Salta,
Argentina y la línea silvestre por el TGRC (Tomato Genetic Resources Center,
Department of Vegetable Crops, University of California, Davis, USA). Para el
proceso se utilizó el método genealógico (Hallauer, 1981). Al final de dicho
proceso, se obtuvieron 18 líneas recombinantes (RIL) (autofecundadas F2:6)
generadas a través de los distintos ciclos por selección antagónica -
divergente de la vida poscosecha (Vp) y el peso (P) de los frutos (Zorzoli
et al., 2000).
El criterio de selección se basó en obtener genotipos con las combinaciones de
ambos caracteres: 1) larga Vp y alto P, 2) corta Vp y bajo P, 3) larga Vp y
bajo P, y 4) corta Vp y alto P. Así se obtuvieron estos 18 cultivares o RIL
discrepantes para el peso, la vida poscosecha y otros atributos que hacen a la
calidad de los frutos (Rodríguez et al., 2006 a, b) (Figura
1).
Estos cultivares fueron caracterizados por RMN- 1H demostrando que S.
pimpinellifolium amplió la variabilidad para la composición química de los
frutos y para algunos metabolitos relacionados con el sabor y componentes
nutricionales (López et al., 2015). Estas RIL son
líneas homocigotas que tienen en su constitución genética fragmentos
recombinados al azar del genoma de ambos progenitores a lo largo de los 12
pares de cromosomas. Estos 18 nuevos cultivares presentan mejores
características de calidad de fruto que el progenitor cultivado del cual
derivan y su tamaño y peso se ajustan al de los llamados tomates tipo cherry.
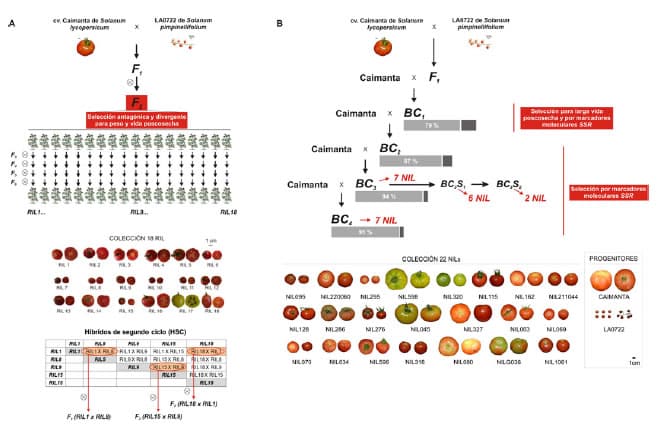
Figura 1. Esquemas de las metodologías de mejoramiento
utilizadas en el Programa de mejoramiento para calidad de fruto de la
Universidad Nacional de Rosario. A) Obtención de RIL (líneas endocriadas
recombinantes) por el método genealógico aplicando selección
antagonistadivergente para el peso y la vida poscosecha de los frutos. B)
Obtención de NIL (líneas casi isogénicas) por el método de las retrocruzas
aplicando selección para larga vida poscosecha y por marcadores moleculares del
tipo SSR.
Esta
población de RIL junto a sus progenitores fueron caracterizados por marcadores
de ADN tales como AFLP (Amplified Fragment Length), SRAP (Sequence Related
Amplified Polymorphism), microsatélites o SSR (Simple Sequence Repeats), InDel
(inserción/ deleción), SNP (Single Nucleotide Polymorphism) y marcadores
funcionales para la forma y el peso de los frutos (Pratta et al., 2011; Mahuad
et al., 2013;
Cambiaso
et al., 2019a;
Cabodevila et al., 2021). También fueron caracterizados por marcadores
proteicos (Gallo et al., 2010). Todos estos
estudios han permitido identificar polimorfismos de ADN y proteínas asociados a
los cambios fenotípicos para la calidad de los frutos.
Obtención de Líneas casi Isogénicas (NIL) por retrocruzas
Inicialmente,
se evaluó la BC1 obtenida de cruzar la F1 (Caimanta x LA0722) por el progenitor
Caimanta. Todos los caracteres evaluados (Vp, P, forma, acidez, contenido en
sólidos solubles, entre otros) mostraron una amplia variación, confirmando la
diversidad genética entre los progenitores y la segregación posterior (Pereira
da Costa et al., 2013).
A partir de estos resultados se comenzó un proceso de detección, validación e
introgresión, en el contexto genético del cultivar Caimanta, de QTL
(Quantitative Trait Loci) asociados a caracteres de calidad de fruto de tomate
aportados por regiones genómicas de la accesión LA0722 de S. pimpinellifolium.
Se continuó con el proceso de introgresión obteniendo las generaciones BC3 y
BC4, seleccionando en cada generación aquellas plantas en las que al menos un
marcador SSR estuviera segregando. Actualmente se dispone de 22 NIL (Near
Isogenic Lines), cada una de las cuales difiere con el progenitor Caimanta en
un segmento cromosómico señalado por 22 marcadores SSR distintos (Figura
1).
Las diferencias observables entre las NIL y el cultivar Caimanta (Di
Giacomo et al., 2020)
se deben exclusivamente a la región introgresada desde la especie donante o
silvestre.
Cruzamientos dialélicos para el estudio de la
variabilidad genética, el efecto recíproco y la heterosis en caracteres de
fruto.
Cinco
de las RIL seleccionadas por sus valores contrastantes fueron evaluadas en un
diseño de cruzamiento dialélico (Híbridos de Segundo Ciclo, HSC) para el P, la
Vp, el contenido en sólidos solubles y la acidez titulable, entre otros. La
existencia de alta variabilidad entre las RIL y la aparición de distintos tipos
de efectos génicos en los HSC, que incluyó el fenómeno de heterosis, demostró
que la variación fenotípica observada responde en gran medida a componentes
genéticos (Marchionni Basté et al., 2010). Estudios recientes
que incluyeron cruzamientos entre tres de nuestras RIL y dos cultivares de
origen estadounidense siguiendo un dialélico completo demostraron que la
calidad y el contenido de metabolitos en los frutos de híbridos de tomate son
afectados por la dirección del cruzamiento y la presencia de heterosis (Fortuny
et al., 2021).
En poblaciones segregantes derivadas de HSC se detectó variabilidad fenotípica
y genética para varios caracteres de calidad de fruto demostrando que es
posible continuar seleccionando genotipos para obtener nuevos cultivares con
características fenotípicas superiores (Cabodevila et al., 2021).
Conservación y divulgación del germoplasma de Tomate
(CodiGo TomATe).
La
biodiversidad presente en las especies silvestres de tomate está aún
subexplotada en los programas de mejoramiento del cultivo especialmente para
enriquecer las bases genéticas con alelos nuevos que mejoren la productividad,
calidad de los frutos y la adaptación del cultivo a distintas condiciones
ambientales. Estudios recientes han detectado que en las especies silvestres de
tomate existen 10 millones más de polimorfismos de una única base (SNP)
comparado con la diversidad presente en la especie cultivada, es decir que la
diversidad en el germoplasma silvestre es 20 veces superior (Aflitos
et al., 2014).
Para conservar y estudiar esta diversidad nuestro grupo está creando una
colección de germoplasma que en la actualidad cuenta con 162 genotipos de
tomate de diferentes especies y orígenes y con variabilidad para
características de fruto. Entre estas tenemos 92 genotipos cultivados de S.
lycopersicum y 70 especies silvestres (7 S. arcanum Peralta, 3 S. chilense, 5
S. corneliomulleri Macbr, 4 S. galapagense Darwin & peralta, 25 S.
habrochaites, 2 S. pennelli, 5 S. peruvianum, 2 S. chmielewskii Rick, Kesicki,
Fobes & Holle, 1 S. neorickii Rick, Kesicki, Fobes & Holle, 3 S.
huaylense Peralta y 13 S. pimpinellifolium). Se multiplican, se cultivan y se
recolectan datos fenotípicos e imágenes con los que se desarrolla el catálogo
de variedades del Banco de Germoplasma de la Cátedra de Genética FCA-UNR. Las
actividades de divulgación del grupo se ilustran en la Figura 2. Además, esta
información junto a otras actividades académicas, de divulgación y
experimentales se difunden a través de la página web www.codigotomate.com.ar.

Figura 2. Actividades para la conservación y
divulgación del germoplasma de tomate. A) Frutos de especies silvestres y
cultivadas de tomate presentes en el catálogo de variedades del Banco de
Germoplasma de la Cátedra de Genética FCA-UNR. La línea blanca horizontal
representa la escala de 1 cm. B) Entrega y trasplante de plantines durante
jornada de extensión en huertas agroecológicas del periurbano de Rosario. C)
Presentación del proyecto CodiGo TomATe en jornadas académicas.
CULTIVARES DESARROLLADOS
Cultivares inscriptos de tomate tipo cherry.
De
las 18 RIL, dos líneas resultaron las más promisorias por sus características
organolépticas, por lo que se han inscripto en el Registro Nacional de
Propiedad de Cultivares (RNPC) del INASE. Ellas son: GEMA FCA y Querubín FCA
(expedientes Nº 79929/12 y Nº 79928/12 publicados en el Boletín Oficial de la
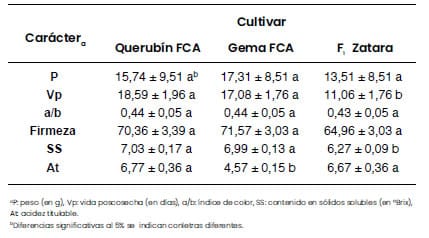
República Argentina Año CXX Número 32.449). En la Figura 3 se muestran frutos
representativos de ambas líneas. Gema FCA y Querubín FCA presentan mayor vida
poscosecha y contenido en azúcares en los frutos que el híbrido comercial
Zatara (Florensa Argentina) de amplia difusión en nuestro país y utilizado en
este ensayo como testigo. Gema FCA, además, produce frutos más ácidos (Tabla 1).

Figura 3. Frutos representativos de los cultivares
tipo cherry Gema FCA y Querubín FCA y tipo redondo Dulcinea FCA y Matusalén FCA
obtenidos en el Programa de Mejoramiento para calidad de fruto de tomate de la
Universidad Nacional de Rosario.. La línea horizontal representa la escala de 1
cm.
Tabla 1. Valores
medios ± error estándar para caracteres de calidad de fruto en los cultivares
tipo cherry Querubín FCA y Gema FCA obtenidos en el Programa de Mejoramiento
Genético FCA/IICAR-CONICET/UNR y el testigo Zatara.
Cultivares experimentales de tomate tipo redondo.
De
las 22 líneas casi Isogénicas (NIL) de tomate, dos líneas resultaron las más
promisorias por lo que se encuentran en evaluación para ser inscriptas en el
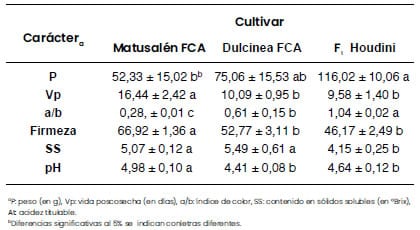
RNPC del INASE. Ellas son: Matusalén FCA y Dulcinea FCA. En la Figura
3 se
muestran frutos representativos de ambas líneas: Matusalén FCA y Dulcinea FCA
producen frutos grandes, pero más chicos que el híbrido comercial Houdini
utilizado como testigo (Florensa Argentina). Matusalén FCA presenta mayor vida
poscosecha y Dulcinea FCA mayor contenido de azúcares en los frutos. Además,
ambas poseen mayor contenido en azúcares y un color más anaranjado que el
testigo (Tabla 2).
Tabla 2. Valores
medios ± error estándar para caracteres de calidad de fruto en los cultivares
tipo redondo Matusalén FCA y Dulcinea FCA obtenidas por el Programa de
Mejoramiento Genético FCA/IICAR-CONICET/UNR y el testigo Houdini.
INCORPORACIÓN DE NUEVAS TECNOLOGÍAS AL PROGRAMA DE MEJORA
Las
innovaciones tecnológicas en el área de la genética y el mejoramiento tales
como los secuenciadores de última generación de genomas y transcriptomas
completos, la edición génica, la adquisición de datos fenotípicos a gran escala
(fenómica), entre otros, generan en la actualidad un crecimiento exponencial
del volumen de los datos que necesitan nuevas metodologías para su análisis
impactando en los programas de mejora. El análisis integral de estos datos no
sólo permite ampliar los conocimientos básicos de los determinantes genéticos
que hacen a la calidad del fruto de tomate, sino que directamente pueden ser la
base para el desarrollo de nuevos cultivares con características mejoradas y
adaptados a los sistemas productivos de nuestro país.
Hemos
recientemente secuenciado el genoma completo del cv. argentino Caimanta y la
entrada LA0722 de S. pimpinellifolium, los que fueron alineados al genoma de
referencia de tomate cv. Heinz 1706 (The Tomato Genome Consortium, 2012). En
base a las listas de polimorfismos se desarrollaron marcadores moleculares para
construir mapas de ligamiento genético y detección de QTL en diferentes
poblaciones del programa (Cambiaso et al., 2019 a,b; Cabodevila et
al., 2021). En cuanto a estudios a nivel transcriptómico, se analizaron los
transcriptos expresados en diferentes estados de madurez en los genotipos
Caimanta y LA0722 por la técnica de cDNA-AFLP (Pereira da Costa et
al., 2018)
y por la técnica de RNA-seq (Cacchiarelli et al.,
2021).
A nivel proteómico hemos caracterizado genotipos uniformes y poblaciones segregantes
por polipéptidos resueltos en 1-DE y 2-DE, para la detección de QTL para
calidad de fruto y la identificación de un polipéptido asociado con cambios en
el pH que mostró homología con una ATPasa cloroplastídica en los pericarpios de
frutos (Rodríguez et al., 2008; Gallo et al., 2010, 2011; Pereira da
Costa et al., 2014, 2017). En cuanto a fenómica, se ha avanzado en el uso del
análisis a tres modos (Análisis Factorial Múltiple y Análisis Procrustes
Generalizado) para caracterizar generaciones segregantes de híbridos de segundo
ciclo y estimar en ellas una heredabilidad multivariada para caracteres de
interés agronómico (Del Medico et al., 2019, 2020). Estas
aproximaciones ómicas permiten identificar genes o familias génicas con roles
fundamentales en la determinación de características organolépticas del fruto
de tomate, conocer su nivel de expresión ante cambios ambientales o de
contextos genéticos y retroalimentan el programa de mejoramiento para obtener
genotipos superiores que satisfagan las demandas de productores y consumidores.
TRANSFERENCIA DE DESARROLLOS TECNOLÓGICOS AL MEDIO
SOCIOPRODUCTIVO
La
horticultura urbana y periurbana es una actividad que aporta múltiples ventajas
territoriales ya que se asocia al mercado de cercanía, ideal para alimentos
perecederos como lo es el tomate, genera puestos de trabajo, y los productores
se convierten en potenciales custodios de los recursos naturales que utilizan
contribuyendo además a preservar los espacios verdes frente al avance de la
urbanización (Mitidieri y Corbino, 2012). Las huertas
urbanas y periurbanas están demandando nuevas metodologías de producción; entre
estas, nuevos cultivares, especialmente de polinización abierta y adaptados a
sistemas de producción agroecológicos. Por esta razón, hemos iniciado un
proyecto que busca una transferencia directa de tecnología de semilla de tomate
hacia el medio socio-productivo y de conocimientos a partir de actividades de
extensión y vinculación con los huerteros urbanos y periurbanos de ciudades
importantes de nuestro país (Figura 2). De esta manera se contribuye a la
soberanía alimentaria favoreciendo el acceso a semillas de cultivares de
tomates desarrollados en instituciones públicas y permitiendo a los productores
multiplicar las semillas para su propio uso. Los genotipos que se transfieren
han sido mejorados para características organolépticas, resistencia a plagas y
enfermedades y algunos de ellos tienen características nutricionales
diferenciales en cuanto al tipo de azúcares, ácidos orgánicos y aminoácidos.
Estas características nutricionales diferenciales, evaluadas en condiciones
experimentales, se confirmarán mediante ensayos en sistemas reales de
producción agroecológica.
BIBLIOGRAFÍA
Aflitos S., Schijlen E., de Jong H.,
de Ridder D., Smit S., Finkers R., Wang J., Zhang G., Li N., Mao L., Bakker F.,
Dirks R., Breit T., Gravendeel B., Huits H., Struss D., Swanson-Wagner R., van
Leeuwen H., van Ham R.C.H.J., Peters S. (2014) Exploring genetic variation in
the tomato (Solanum section Lycopersicon) clade by whole-genome sequencing. Plant
J. 80: 136-148.
Blanca J., Sanchez-Matarredona D.,
Ziarsolo P., Montero-Pau J., van der Knaap E., Díez M.J., Cañizares J. (2021) Haplotype
analyses reveal novel insights into tomato history and domestication to show
long-distance migrations and latitudinal adaptations. BioRxiv. 1-41.
Brummell D.A. and Harpster M.H. (2001)
Cell wall metabolism in fruit softening and quality and its manipulation in
transgenic plants. Plant Mol. Biol. 47(1-2): 311-339.
Cabodevila V.G., Cambiaso V., Rodríguez
G.R., Picardi L.A., Pratta G.R., Capel C., Lozano R., Capel J. (2021) A
segregating population from a tomato second cycle hybrid allows the
identification of novel QTL for fruit quality traits. Euphytica. 217:6.
Cabodevila V.G., Picardi L.A., Pratta
G.R. (2017) A multivariate approach to explore the genetic variability in the
F2 segregating population of a tomato second cycle hybrid. BAG. J. Basic Appl.
Genet. 28:7-18.
Cacchiarelli P., Arce D.P., Tapia
E., Pratta G.R. (2021) Structural and functional analysis of two sHSP
subfamilies in tomato ripening. Plant Gene. 27:100297.
Cambiaso V., Gimenez M.D., Pereira
da Costa J.H., Vazquez D.V., Picardi L.A., Pratta G.R., Rodríguez G.R. (2019a) Selected genome regions for fruit weight
and shelf life in tomato RILs discernible by markers based on genomic sequence
information. Breed. Sci. 69(3): 447-454.
Cambiaso V., Pratta G.R., Pereira
da Costa J.H., Zorzoli R., Francis D.M., Rodríguez G.R. (2019b) Whole genome
re-sequencing analysis of two tomato genotypes for polymorphism insight in
cloned genes and a genetic map construction. Sci. Hortic. 247: 58-66.
Cattáneo A.R., McCarthy A.N., Feingold
S.E. (2020) Evidence of genetic diversity within Solanum Lycopersicum L.
‘Platense’ landrace and identification of various subpopulations. Genet.
Resour. Crop Ev. 67(8): 2057-2069.
Del Medico A.P., Cabodevila V.G.,
Vitelleschi M.S., Pratta G.R. (2019) Multivariate estimate of heritability for
quality traits in tomatoes by the multiple factor analysis. Pesqui. Agropecu.
Bras. 54: e00064.
Del Medico A.P., Cabodevila V.G.,
Vitelleschi M.S., Pratta G.R. (2020) Characterization of tomato generations
according to a three-way data analysis. Bragantia. 79(1): 8-18.
Di Giacomo M., Luciani M.D., Cambiaso
V., Zorzoli R., Rodríguez G.R., Pereira da Costa J.H. (2020) Tomato near
isogenic lines to unravel the genetic diversity of S. pimpinellifolium LA0722
for fruit quality and shelf life breeding. Euphytica
216(8): 126.
FAO STAT (2021) http://www.fao.org/home/es/
(accessed June 2021).
Fortuny A.P., Bueno R.A., Pereira
da Costa J.H., Zanor M.I., Rodríguez G.R. (2021) Tomato fruit quality traits
and metabolite content are affected by reciprocal crosses and heterosis. J.
Exp. Bot. https://doi.org/10.1093/jxb/erab222.
Gallardo G.S. (2012) Desarrollo
institucional y política científica: el caso de la producción nacional de
semilla hortícola. Tesis para optar al grado académico de Magister en la
Maestría en Gestión de la Ciencia, Tecnología e Innovación de la Universidad
Nacional de General Sarmiento, Buenos Aires, Argentina.
Gallo M., Picardi L.A., Rodríguez
G.R., Pratta G.R., Zorzoli R. (2010) Proteómica de la madurez del tomate:
Identificación de dos estados de madurez del fruto por perfiles proteicos
totales del pericarpio en RILs de tomate. Rev. FCA UNCuyo. 42(2): 119-133.
Gallo M., Zorzoli R., Rodríguez G.R.,
Pratta G.R. (2011)
Ligamiento genético entre variables asociadas a calidad del fruto de tomate y
polipéptidos expresados en dos estados de madurez. Rev. FCA UNCuyo. 43(2):145-156.
Hallauer, A.R. (1981) Selection
and breeding methods. In: K.J. Frey (Ed.). Plant breeding II. Iowa State Univ.
Press, Ames, pp. 3-55.
Ishii T., Araki M. (2016) Consumer
acceptance of food crops developed by genome editing. Plant Cell Rep. 35(7): 1507-1518.
López M.G., Zanor M.I., Pratta G.R.,
Stegmayer G., Boggio S.B., Conte M., Bermúdez L., Coluccio Leskow C., Rodríguez
G.R., Picardi L.A., Zorzoli R., Fernie A.R., Milone D., Asís R., Valle E.M., Carrari
F. (2015) Metabolic analyses of interspecific tomato recombinant inbred lines
for fruit quality improvement. Metabolomics. 11: 1416-1431.
Mahuad S.L., Pratta G.R., Rodríguez
G.R., Zorzoli R., Picardi L.A. (2013) Preservation of Solanum pimpinellifolium
genomic fragments in recombinant genotypes improved the fruit quality of tomato.
J. Genet. 92(2): 195-203.
Manning K., Tör M., Poole M., Hong
Y., Thompson A.J., King G.J., Giovannoni J.J., Seymour G.B. (2006) A naturally
occurring epigenetic mutation in a gene encoding an SBP-box transcription
factor inhibits tomato fruit ripening. Nat. Genet. 38(8): 948-952.
Marchionni Basté E., Liberatti D.R.,
Mahuad S.L., Rodríguez G.R., Pratta G.R., Zorzoli R., Picardi L.A. (2010) Diallel
analysis for fruit traits among tomato recombinant inbred lines derived from an
interspecific cross Solanum lycopersicum x S. pimpinellifolium. J. Appl. Hortic.
12(1): 21-25.
Meli V.S., Ghosh S., Prabha T.N.,
Chakraborty N., Chakraborty S., Datta A. (2010) Enhancement of fruit shelf life
by suppressing N-glycan processing enzymes. Proc. Natl. Acad. Sci. U.S.A. 107(6):
2413-2418.
Ministerio de Agricultura,
Ganadería y Pesca (2020) La Producción de Tomate en Argentina. https://www.argentina.gob.ar/sites/default/files/producciontomate-argentina-diciembre-2020.pdf
(accessed June 2021).
Mitidieri M.S., Corbino G.B. (2012)
Manual de horticultura periurbana. Instituto Nacional de Tecnología Agropecuaria,
San Pedro, Buenos Aires, Argentina.
Pereira da Costa J.H., Rodríguez G.R.,
Picardi L.A., Zorzoli R., Pratta G.R. (2018) Genome-wide expression analysis at
three fruit ripening stages for tomato genotypes differing in fruit shelf life. Sci. Hortic . 229: 125-131.
Pereira da Costa J.H., Rodríguez G.R.,
Pratta G.R., Picardi L.A., Zorzoli R. (2013) QTL detection for fruit shelf life
and quality traits across segregating populations of tomato. Sci. Hortic. 156: 47-53.
Pereira da Costa J.H., Rodríguez G.R.,
Pratta G.R., Picardi L.A., Zorzoli R. (2014) Pericarp polypeptides and SRAP
markers associated with fruit quality traits in an interspecific tomato
backcross. Genet. Mol. Res. 13(2): 2539-2547.
Pereira da Costa J.H., Vega T.A.,
Pratta G.R., Picardi L.A., Zorzoli R., Rodríguez G.R. (2017) A 54-kDa
polypeptide identified by 2D-PAGE and bulked segregant analysis underlies differences for pH
values in tomato fruit. Acta Physiol. Plant. 39: 78.
Pratta G., Zorzoli R., Picardi L.A.
(2000) Interacciones
genéticas entre germoplasma silvestre y cultivado de Lycopersicon spp. con
efectos sobre la calidad del fruto de tomate. Plant Genet. Resour. Newsl. 124: 7-12.
Pratta G.R., Rodríguez G.R., Zorzoli
R., Valle E.M., Picardi L.A. (2011) Phenotypic and molecular characterization
of selected tomato recombinant inbred lines derived from the cross Solanum
lycopersicum x S. pimpinellifolium. J. Genet. 90(2):
229-237.
Rasmussen W.D. (1968) Advances in
American Agriculture: The Mechanical Tomato Harvester as a Case Study. Technol.
Cult. 9(4): 531-543.
Razifard H., Ramos A., Della
Valle A.L., Bodary C., Goetz E., Manser E.J., Li X., Zhang L., Visa S., Tieman D.,
van der Knaap E., Caicedo A.L. (2020) Genomic evidence for complex
domestication history of the cultivated tomato in Latin America. Mol. Biol.
Evol. 37(4): 1118-1132.
Rodríguez G.R., Pratta G.R., Liberatti
D.R., Zorzoli R., Picardi L.A. (2010) Inheritance of shelf life and other
quality traits of tomato fruit estimated from F1’s, F2’s and backcross
generations derived from standard cultivar, nor homozygote and wild cherry
tomato. Euphytica. 176(1): 137- 147.
Rodríguez G.R., Pratta G.R., Zorzoli
R., Picardi L.A. (2006a)
Evaluación de caracteres de planta y frutos en líneas recombinantes
autofecundadas de tomate obtenidos por cruzamiento entre Lycopersicom
esculentum y L. pimpinellifolium. Cienc.
Investig. Agrar. 33(2): 133-141.
Rodríguez G.R., Pratta G.R., Zorzoli
R., Picardi L.A. (2006b) Recombinant lines obtained from an interspecific cross
between lycopersicon species selected by fruit weight and fruit shelf life. J.
Am. Soc. Hortic. Sci. 131(5): 651-656.
Rodríguez G.R., Sequin L., Pratta
G.R., Zorzoli R., Picardi L.A. (2008) Protein profiling in F1 and F2
generations of two tomato genotypes differing in ripening time. Biol. Plant. 52(3):
548- 552.
Schmidt S.M., Belisle M., Frommer
W.B. (2020) The evolving landscape around genome editing in agriculture. EMBO
Reports. 21(6): 19-22.
Schouten H.J., Tikunov Y., Verkerke
W., Finkers R., Bovy A., Bai Y., Visser R.G.F. (2019) Breeding Has Increased
the Diversity of Cultivated Tomato in The Netherlands. Front. Plant Sci. 10: 1-12.
Sim S.C., Robbins M.D., Chilcott C.,
Zhu T., Francis D.M. (2009) Oligonucleotide array discovery of polymorphisms in
cultivated tomato (Solanum lycopersicum L.) reveals patterns of SNP variation
associated with breeding. BMC Genomics. 10: 466.
Sim S.C., van Deynze A., Stoffel K.,
Douches D.S., Zarka D., Ganal M.W., Chetelat R.T., Hutton S.F., Scott J.W., Gardner
R.G., Panthee D.R., Mutschler M., Myers J.R., Francis D.M. (2012) High-density
SNP genotyping of tomato (Solanum lycopersicum L.) reveals patterns of genetic
variation due to breeding. PloS One. 7(9): e45520.
Spooner D.M., Peralta I.E., Knapp
S. (2005) Comparison of AFLPs with other markers for phylogenetic inference in
wild tomatoes [Solanum L. section Lycopersicon (Mill.) Wettst.].
Taxon. 54(1): 43-61.
The Tomato Genome Consortium. (2012).
The tomato genome sequence provides insights into fleshy fruit evolution. Nature.
485(7400): 635-641.
Uluisik S., Chapman N.H., Smith R.,
Poole M., Adams G., Gillis R.B., Besong T.M.D., Sheldon J., Stiegelmeyer S., Perez
L., Samsulrizal N., Wang D., Fisk I.D., Yang N., Baxter C., Rickett D., Fray R.,
Blanco- Ulate B., Powell A.L.T., Seymour G.B. (2016) Genetic improvement of
tomato by targeted control of fruit softening. Nat. Biotechnol. 1(9): 1-11.
Vrebalov J. (2002) A MADS-Box
Gene Necessary for Fruit Ripening at the Tomato Ripening-Inhibitor (Rin) Locus.
Science. 296(5566): 343-346.
Wang D., Samsulrizal N., Yan C., Allcock
N.S., Craigon J., Blanco-Ulate B., Ortega-Salazar I., Marcus S.E., Bagheri H.M.,
Perez-Fons L., Fraser P.D., Foster T., Fray R.G., Knox J.P., Seymour G.B. (2019)
Characterisation of CRISPR mutants targeting genes modulating pectin
degradation in ripening tomato. Plant Physiol. 179: 544-557.
Warnock S.J. (1991) Natural
Habitats of Lycopersicon Species. HortScience.
26(5): 466-471.
Yang Y., Zhu G., Li R., Yan S., Fu
D., Zhu B., Tian H., Luo Y., Zhu H. (2017) The RNA editing factor SLORRM4 is
required for normal fruit ripening in tomato. Plant
Physiol. 175(4): 1690-1702.
Yu Q.H., Wang B., Li N., Tang Y.,
Yang S., Yang T., Xu J., Guo C., Yan P., Wang Q., Asmutola P. (2017) CRISPR/Cas9-
induced Targeted Mutagenesis and Gene Replacement to Generate Long-shelf Life
Tomato Lines. Sci. Rep. 7(1): 1-9.
Zorzoli R., Pratta G.R., Picardi L.A.
(1998) Efecto
de los mutantes nor y rin y de genes silvestres sobre características del fruto
en Lycopersicon. Mendeliana. 13: 12-19.
Zorzoli R., Pratta G.R., Picardi L.A.
(2000) Variabilidad
para la vida postcosecha y el peso de los frutos en tomate para familias F3 de
un híbrido interespecífico. Pesqui. Agropecu. Bras. 35: 2423-2427.



