Vol. XXXIII Issue 2
Article 4
DOI: 10.35407/bag.2022.33.01.04
ARTÍCULOS
ORIGINALES
Detección clonal de
cepas parentales de streptococcus agalactiae Lehmann y Neumann por
amplificación aleatoria de ADN polimórfico
Clonal detection of streptococcus agalactiae Lehmann and Neumann
parental strains by random amplification of polymorphic DNA
Cortese
I.J. 1,2
Novosak
M.G. 1,2
Oviedo P.N. 2
Cannistraci Giolito R.E. 3
Laczeski M.E.1,2 *
1 Laboratorio de
Biotecnología Molecular, Instituto de Biotecnología Misiones “Dra. María Ebe
Reca” (InBioMis), CONICET, Facultad de Ciencias Exactas, Químicas y
Naturales/FCEQyN, Universidad Nacional de Misiones/UNaM, Ruta 12 Km 7,5 (CP 3304),
Posadas, Misiones, Argentina.
2 Cátedra de
Bacteriología, Dpto. de Microbiología, Facultad de Ciencias Exactas, Químicas y
Naturales/FCEQyN, Universidad Nacional de Misiones/UNaM, Avenida Mariano Moreno
1375 (CP 3300), Posadas, Misiones, Argentina.
3 Cátedra de
Bacteriología y Virología Médicas, Facultad de Medicina, Universidad Nacional
de Córdoba. Escuela Práctica, Santa Rosa 1095 (CP 5000), Córdoba,
Argentina.
*
Corresponding author:
Margarita Ester Laczeski melaczeski@fceqyn.unam.edu.ar ORCID
0000-0002-8303-7874
RESUMEN
Streptococcus
agalactiae (SGB)
produce infecciones invasivas en neonatos siendo la transmisión materna la más
frecuente. Estudios epidemiológicos utilizan técnicas moleculares que evalúan
la diversidad genética, entre ellas la de amplificación aleatoria de ADN
polimórfico (RAPD) que resulta ser accesible, sensible y utiliza cebadores
arbitrarios para amplificar segmentos polimórficos de ADN mediante PCR. El
objetivo fue determinar la relación clonal entre aislamientos de SGB
recuperados de madres y sus respectivos recién nacidos. Se estudiaron por RAPD
cuatro parejas de aislamientos de SGB obtenidos de hisopados vagino-rectales de
madres y de hemocultivos de sus neonatos. Se utilizaron los cebadores OPS11, OPB17
y OPB18 para seleccionar uno con capacidad de discriminar entre cepas no
relacionadas genéticamente. Se utilizó la fórmula de Hunter-Gaston que
establece el índice de discriminación (D), cuando D>0,90 se considera que
los aislamientos pertenecen a clones diferentes. Los perfiles de amplificación
para los ocho aislamientos, empleando independientemente cada cebador,
permitieron calcular un D=1 para OPS11, y D=0,84 para OPB17 y OPB18. Por lo
tanto, OPS11 fue seleccionado para el estudio de la relación clonal de los
aislamientos, encontrándose perfiles de amplificación similares por RAPD para
cada par de cepas madre-recién nacido. Se observaron diferentes perfiles de
amplificación entre los pares de cepas madre-recién nacido, lo que revela la
discriminación entre cepas no relacionadas, resultados confirmados por
electroforesis en campo pulsante (PFGE). Estos resultados indican transmisión
vertical para cada caso estudiado y robustez del cebador OPS11. Se encontraron
condiciones apropiadas del ensayo de RAPD, lo que es útil para estudios
epidemiológicos.
Palabras
clave:
Streptococcus agalactiae, Enfermedad
neonatal, Epidemiología molecular, Técnica RAPD, Transmisión
vertical
ABSTRACT
Streptococcus agalactiae (GBS) causes invasive infections in newborns, being the most frequent the
maternal transmission. Epidemiological studies use molecular techniques that
assess genetic diversity, including random amplification of polymorphic DNA
(RAPD) that is found to be accessible, sensitive and uses arbitrary primers to
amplify polymorphic segments of DNA by PCR. The objective was to determine the
clonal relationship between GBS strains recovered from mothers and their
respective newborns. Four pairs of GBS isolates obtained from vaginal-rectal swabs
of mothers and blood cultures of their newborns were studied with RAPD. Primers
OPS11, OPB17 and OPB18 were used to select one with the ability to discriminate
between non-genetically related strains. The Hunter-Gaston formula that
establishes the discrimination index (D) was used; when D>0.90, it is
considered that the isolates belong to different clones. The amplification profiles
for the eight isolates, using each primer independently, allowed to calculate a
D=1 for OPS11, and D=0.84 for OPB17 and OPB18. Therefore, OPS11 was selected
for the study of the clonal relationship of the isolates, and similar amplification
profiles were found by RAPD for each mother-newborn pair of GBS isolates.
Different amplification profiles were observed between pairs of mother-newborn
strains, which reveals the discrimination between unrelated strains, confirmed
by pulsating field electrophoresis (PFGE). These results indicated vertical
transmission for each studied case and robustness of the OPS11 primer.
Appropriate conditions of the RAPD trial were found, which is useful for
epidemiological studies.
Key words: Streptococcus agalactiae, Neonatal
disease, Molecular epidemiology, RAPD technique, Vertical
transmission
Received: 03/29/2021
Revised version received: 07/13/2021
Accepted: 09/22/2021
General
Editor:
Elsa Camadro
INTRODUCCIÓN
Streptococcus
agalactiae (Streptococcus
del grupo B, SGB) es un microorganismo que coloniza el tracto gastrointestinal
y genitourinario humano (Barcaite et al., 2014). Su colonización puede
ser transitoria, intermitente o crónica, y los factores que influyen en su persistencia
aún son desconocidos (Patras et al., 2015; Sarrión-Sos
et al., 2018).
La
enfermedad invasiva por SGB se identificó por primera vez en adultos y neonatos
en la década de 1960 (Raabe y Shane, 2019). La vía de
transmisión al feto o al recién nacido ocurre a partir de la colonización recto-vaginal
materna (Russell et al., 2017). En mujeres embarazadas
colonizadas, las bacterias pueden ascender desde el tracto genitourinario hacia
el útero o la vejiga. En el útero, pueden afectar las membranas fetales y
causar corioamnionitis, lo que puede provocar parto prematuro, aborto
espontáneo o la muerte fetal intrauterina. Alternativamente, la bacteria puede invadir
el tracto respiratorio del recién nacido mediante la aspiración del líquido
amniótico o por su pasaje por el canal de parto y causar neumonía. También
puede destruir el revestimiento alveolar utilizando factores de virulencia y
llegar al torrente sanguíneo, desencadenando bacteriemia y sepsis. Una vez en
sangre, las bacterias pueden cruzar la barrera hematoencefálica y migrar al líquido
cefalorraquídeo (LCR) produciendo meningitis (Hanna y Noor, 2020). Las tasas de
transmisión del SGB madre-recién nacido varían en todo el mundo, sin embargo,
los informes describen tasas de alrededor de 0,53 por 1.000 nacidos vivos (Do
Nascimento et al., 2019). Se describieron dos formas de enfermedad
invasiva por SGB en niños en función de la edad de presentación: la enfermedad
de inicio temprano (early onset disease; EOD) que ocurre durante los
primeros seis días de vida, y la enfermedad de inicio tardío (late onset
disease; LOD) que se desarrolla entre los siete y 90 días después del nacimiento,
se manifiesta con meningitis y bacteriemia y es de transmisión materna o
intranosocomial (Manrique Martín et al., 2020).
La
EOD, la más severa y de mayor importancia clínica, surge de la transmisión
vertical de una madre colonizada a su recién nacido durante o inmediatamente
antes del nacimiento, con signos clínicos que ocurren dentro de las 24-48 h en
más del 90% de los casos (Collin et al., 2019). Los factores de riesgo
para la EOD incluyen colonización vaginal o rectal materna, bacteriuria por SGB
durante el embarazo, trabajo de parto prolongado, rotura prematura de
membranas, bajo peso al nacer, prematuridad, fiebre intraparto y enfermedad
sistémica materna por SGB (Nanayakkara et al.,
2018;
Raabe
y Shane, 2019).
Por otro lado, las manifestaciones típicas de la EOD incluyen dificultad
respiratoria como apnea o taquipnea, respiración entrecortada, cianosis,
letargo, mala alimentación, distensión abdominal, palidez, ictericia,
taquicardia e hipotensión. La bacteriemia es la forma más común de EOD
producida por SGB, manifestada en el 80% de los casos, mientras que la neumonía
y meningitis son menos comunes y representan el 15% y 5% a 10%, respectivamente
(Raabe y Shane, 2019; Hanna y Noor, 2020). La gravedad de la EOD está
determinada en gran medida por factores de virulencia necesarios para la interacción
célula huésped-bacteria (Herbert et al., 2004); uno de ellos, la
cápsula polisacárida, permite la clasificación de SGB en los serotipos Ia, Ib,
II a IX (Hanna y Noor, 2020).
Algunos
serotipos se asocian con clones más virulentos y, por lo tanto, con una
propensión a la enfermedad invasiva por SGB, por ejemplo, el III, que se asocia
con frecuencia con el complejo clonal hipervirulento y representa el 43% de la
EOD y el 73% de la LOD, y el IV, que causa el 97% de las infecciones severas en
neonatos (Raabe y Shane, 2019; Russell
et al., 2017).
A partir de 1996 el Centro para el Control y la Prevención de Enfermedades
(CDC, 1996; CDC, 2002; CDC, 2010) publicó directrices para la prevención de la
enfermedad perinatal, a partir de la búsqueda de SGB en toda mujer embarazada
entre 35 y 37 semanas de gestación. Teniendo en cuenta la importancia de implementar
acciones y decisiones de salud pública, en nuestro país, desde abril del año
2008, se encuentra vigente la Ley Nacional Nº 26.369 (2008) que establece la
obligatoriedad de la búsqueda de portación de SGB en toda mujer con edad
gestacional entre 35 y 37 semanas, presenten o no condiciones de riesgo.
En
las últimas tres décadas se han desarrollado técnicas moleculares orientadas al
estudio de la diversidad genética entre microorganismos estrechamente
relacionados, entre ellas polimorfismos de longitud de fragmentos de
restricción (RFLP) (Chatellier et al., 1996), electroforesis en
gel de campo pulsado (PFGE) (Manning, 2003), electroforesis enzimática
multilocus (Quentin et al., 1995), ribotipado (Huet
et al., 1993)
y amplificación aleatoria de ADN polimórfico (RAPD) (Toresani
et al., 2001).
Si bien se ha demostrado que PFGE es una técnica precisa y fiable, utilizada
como método estándar en laboratorios de mediana y alta complejidad (Åberg
et al., 2019),
su aplicación es compleja, costosa y requiere de mucho tiempo en comparación
con la facilidad y velocidad de ejecución de las técnicas basadas en la
reacción en cadena de la polimerasa (PCR). En este sentido la técnica RAPD es accesible
y sensible, por lo que su aplicación en estudios de variabilidad es
especialmente útil para realizar análisis intraespecíficos. Esta técnica se
realiza con pequeñas cantidades de ADN y con cebadores sintéticos cortos con
una secuencia al azar de aproximadamente 9-10 bases de longitud, cuya selección
es un paso crítico. Estos marcadores tienen la ventaja de amplificar regiones
del genoma que pueden ser transcriptas como así también regiones no
codificantes (Rolim et al., 2011). La amplificación
de fragmentos se produce debido a las diferencias en la distancia entre los
sitios de unión del cebador produciendo perfiles de bandas específicos para una
cepa determinada (Idil y Bilkay, 2014).
En
este contexto, si bien la pesquisa de colonización materna y la instauración de
la profilaxis intraparto (PIP) con antimicrobianos a toda embarazada colonizada
es obligatoria, la mortalidad del recién nacido es aún elevada, principalmente
en partos pre-término o con factores de riesgo subyacentes. Por ello, el
desafío actual está orientado a desarrollar nuevos métodos de diagnóstico y
nuevas estrategias de prevención que incluyan el desarrollo de vacunas a fin de
prevenir la incidencia de este microorganismo en el recién nacido (Chen
et al., 2013;
Szymusik
et al., 2014).
Además, resulta importante para la región y el país, realizar estudios
epidemiológicos ligados a la portación materna de SGB y su transmisión al
recién nacido, y a la posible transmisión horizontal en centros de salud. Por ello,
el objetivo de esta investigación fue determinar la relación clonal entre
aislamientos de SGB recuperados de madres colonizadas y sus respectivos recién
nacidos de dos hospitales argentinos por la técnica RAPD.
MATERIALES Y MÉTODOS
Bacterias
Se
trabajó con cuatro aislamientos de SGB recuperados de mujeres luego del parto
(estas pacientes habían llegado a los centros de salud sin realización previa de
búsqueda de portación materna de SGB durante el embarazo) y cuatro aislamientos
recuperados de sus respectivos recién nacidos con enfermedad invasiva por SGB,
entre los años 2011 y 2016. Tres pares de aislamientos madre-hijo fueron del
Hospital Escuela de Agudos “Dr. Ramón Madariaga” de Posadas, Misiones y el par
restante del Hospital Materno Provincial “Dr. Raul F. Lucini” de Córdoba,
Córdoba. Los seis aislamientos de Posadas se identificaron como E4669 madre y
O4663 recién nacido; E2949 madre y O2963 recién nacido; E6149 madre y O6150
recién nacido (además, para la técnica de PCR se incluyó el aislamiento O6151
recién nacido recuperado de LCR del mismo paciente) y los dos de Córdoba como 6
madre y 7 recién nacido. Las muestras maternas se tomaron con hisopo estéril
del tercio anterior de vagina y de la zona anorectal. Se conservaron en medio
de transporte Stuart (Venturi Transystem Stuart, Copan, Italia S.p.A) hasta la siembra
en 1-2 mL de caldo Todd-Hewitt suplementado con colistina (10 μg mL-1,
Britania, Argentina) y ácido nalidíxico (15 μg mL-1, Britania, Argentina)
y se incubaron a 37° C durante 24 h. Luego se sembraron con técnica para
aislamiento en placas de agar con sangre ovina al 5% (Rafaela, Argentina). Las
placas se incubaron en atmósfera microaeróbica a 37° C durante 24 h. La
identificación bacteriana se realizó mediante pruebas bioquímicas
convencionales y serología de grupo (aglutinación con partículas de látex,
Phadebact Strep B Test-ETC International-Bactus AB, Suecia). El serotipo se
determinó por técnica de aglutinación Statens Serum Institute (Strep-B. Latex,
Copenhague, Dinamarca). Ambas técnicas se realizaron siguiendo las recomendaciones
del fabricante. De los recién nacidos se tomaron muestras de hemocultivos y LCR
siguiendo las recomendaciones internacionales. La siembra e identificación
bioquímica de los aislamientos neonatales de SGB se realizó con las mismas
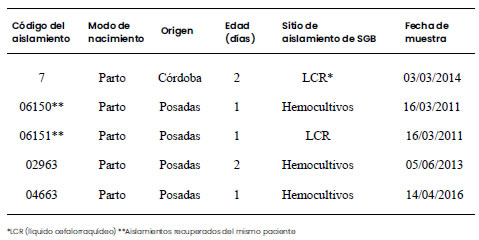
técnicas utilizadas para los aislamientos maternos. En la Tabla 1 se detallan
las características de los aislamientos de SGB recuperados de los recién nacidos.
Todos los aislamientos se conservaron en leche descremada al 20% a -80° C en el
Cepario de la Cátedra de Bacteriología, Facultad de Ciencias Exactas, Químicas y
Naturales, Universidad Nacional de Misiones, hasta ser utilizados en esta
investigación.
Tabla 1. Datos de los
aislamientos de Streptococcus agalactiae recuperados de recién nacidos
con enfermedad invasiva de inicio temprano. Posadas-Córdoba, 2011-2016
Extracción de ADN
Los
aislamientos de SGB se cultivaron en caldo Todd- Hewitt (Britania, Argentina) y
se incubaron a 37° C durante 24 h. La extracción de ADN bacteriano se realizó con
el protocolo de Sambrook modificado (Cariaga Martinez y Zapata, 2007).
Selección de
cebadores
Se
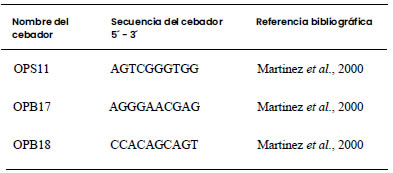
realizó un análisis comparativo de los patrones de bandas generados para los
aislamientos a partir de la técnica RAPD utilizando tres cebadores previamente diseñados
para SGB (Martinez et al., 2000) (Tabla 2). Se
evaluó la utilidad de cada cebador para detectar aislamientos genéticamente no
relacionados con el Índice de Simpson (D), calculado a partir de la siguiente fórmula
(Hunter
y Gaston, 1998).
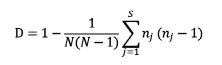
Donde
N es el número total de los aislamientos en la población de la muestra, S es el
número total de patrones de RAPD obtenidos y nj es el número de aislamientos pertenecientes
al tipo j-ésimo. Un índice superior a 0,90 permite la interpretación de
resultados de tipificación fiables (Hunter y Gaston, 1998; Martinez
et al., 2000;
Shadbad
et al., 2020).
Tabla 2. Secuencias
nucleotídicas de los cebadores utilizados para la técnica RAPD.
Generación de
patrones RAPD
La
reacción de amplificación se realizó en un volumen final de 50 μL
compuesto por solución tampón para PCR 1X (10X: 100 mM Tris-HCl pH 9, 500 mM
KCl, 1% Triton® X-100), 100 mM dNTPs, 0,4 μM de cebador (Tabla
2), 2,5 mM MgCl2, 50 ng μL-1 de ADN molde y 2,5 U de ADN polimerasa Taq
(Inbio Highway, Argentina).
Se
utilizó un solo cebador en cada reacción de PCR. Las amplificaciones se
realizaron en un termociclador Multigene TM II (Labnet internacional Inc., EE.
UU.). El perfil de ciclado consistió en: pre-desnaturalización a 94° C durante
5 min, 40 ciclos de amplificación (desnaturalización a 94° C durante 1 min,
hibridación a 36° C durante 1 min, extensión a 72° C durante 1 min) y extensión
a 72° C durante 5 min. Los productos de amplificación se revelaron en gel de
agarosa al 1,2% (p/v) teñido con GelRed® (Sigma-Aldrich, Alemania). La
electroforesis se realizó en una cuba electroforética (Subsistema de
electroforesis 70 Labnet International) a 100 V durante 3 h y las bandas se
visualizaron usando un transiluminador UV (Modelo MUV 21-312-220).
Análisis de patrones
de bandas obtenidos por RAPD y construcción del Dendrograma
En
base a la distancia de migración, el tamaño de las diferentes bandas fue medido
y extrapolado al correspondiente marcador de peso molecular mediante el
programa Graphpad Prism.
Los
dendrogramas se generaron con el programa estadístico para Excel, XLSTAT utilizando
el método de grupos de pares no ponderados con media aritmética (UPGMA). Se
consideró que los aislamientos que tenían una similitud >75% en el índice de
Dice con una tolerancia de 3,5 tenían el mismo origen, lo que indicaría una
posible transmisión entre la madre y el recién nacido.
Electroforesis en gel
de campo pulsante
Los
pares de cepas madre-recién nacido fueron enviadas al Servicio de
Antimicrobianos de la Administración Nacional de Laboratorios e Institutos de
Salud (ANLIS) “Dr. Carlos Malbrán” para evaluar su relación clonal mediante
PFGE. De acuerdo con el informe recibido, el ADN total se digirió con las
enzimas de restricción SmaI y ApaI y se trabajó de acuerdo con el
protocolo propuesto por Faccone et al. (2010) utilizando un
equipo CHEFDRIII.
RESULTADOS
Se
detectó el serotipo III en tres pares de aislamientos madre-recién nacido, y el
serotipo Ia en uno de ellos.
El
cebador OPS11 mostró un perfil de amplificación nítido y un valor D igual a 1,
mientras que los cebadores OPB17 y OPB18 mostraron patrones de bandas no concluyentes
y un valor D de 0,84. En la Figura 1 se muestran los perfiles de bandas
obtenidos a partir de la técnica RAPD utilizando los tres cebadores. En base a
los patrones de bandas observados y a los valores obtenidos para D, se
seleccionó el cebador OPS11 para el análisis de los pares de cepas madre-recién
nacido, y para su posterior comparación con los resultados generados por la
técnica PFGE. El cebador OPS11 permitió caracterizar individualmente a cada
cepa estudiada. El análisis genómico de los aislados de SGB reveló cuatro perfiles
de bandas diferentes indicando la presencia de cuatro clones, lo que demuestra
una relación entre el aislamiento materno y la infección del recién nacido en
los cuatro casos. Por otro lado, no se evidenció una relación epidemiológica
entre las cepas de diferentes pares de madre-recién nacido, a pesar de
pertenecer al mismo serotipo (Figura 2).
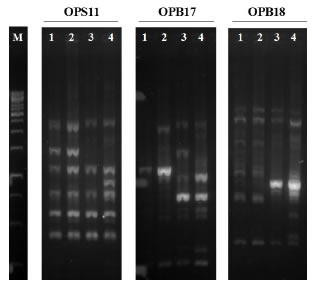
Figura 1. Perfiles de bandas
electroforéticas obtenidos por la técnica RAPD utilizando los cebadores OPS11,
OPB17 y OPB18.
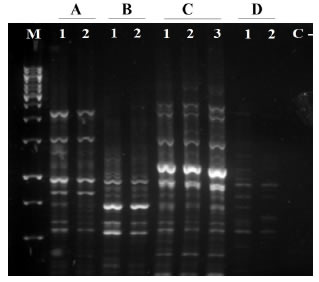
Figura 2. Perfiles de bandas
electroforéticas obtenidos por la técnica RAPD utilizando el cebador OPS11,
comparación entre los aislamientos de Streptococcus agalactiae obtenidos
de madre-recién nacido: (A) E4669 madre y O4663 recién nacido; (B) E2949 madre
y O2963 recién nacido; (C) E6149 madre y O6150 recién nacido; O6151 recién
nacido y (D) 6 madre y 7 recién nacido.
El
informe de PFGE del Servicio de Antimicrobianos de la Administración Nacional
de Laboratorios y Servicios de Salud (ANLIS) “Dr. Carlos Malbrán”, fue el siguiente:
La restricción con ApaI, sólo permitió tipificar las cepas 6 madre y 7
recién nacido que dieron perfiles indistinguibles entre sí. Los seis
aislamientos restantes resultaron no con ApaI en tres oportunidades. Usando
SmaI, los aislamientos 6 madre y 7 recién nacido presentaron un perfil
de restricción similar con dos bandas de diferencia, lo que implicaría que
corresponden a subtipos de un mismo clon. Los seis aislamientos restantes E4669
madre y O4663 recién nacido; E2949 madre y O2963 recién nacido; E6149 madre y
O6150 recién nacido, que no pudieron tipificarse con ApaI, al hacer la
restricción con SmaI se pudieron diferenciar en tres tipos clonales.
Cada uno de estos tipos clonales corresponde a una dupla madre e hijo,
indistinguibles entre sí. Los resultados obtenidos por PFGE permiten determinar
que en todos los casos los pares de aislamientos madre-recién nacido estuvieron
genéticamente relacionados entre sí, respaldando los resultados generados
previamente con la técnica RAPD utilizando el cebador OPS1. 1
DISCUSIÓN
La
diversidad genética entre cepas de SGB estrechamente relacionadas ha sido
estudiada a partir de diferentes técnicas moleculares. Algunas metodologías como
PFGE implican pasos que consumen mucho tiempo, reactivos costosos y equipos
sofisticados, por lo que está restringida a laboratorios de referencia donde es
aplicada como técnica estándar (Hansen et al.,
2004).
Sin embargo la técnica RAPD, entre otras, ha sido utilizada en los últimos 30
años para determinar la relación entre los aislamientos de SGB (Shadbad
et al., 2020;
Taniyama
et al., 2020).
Se ha aplicado ampliamente para el análisis epidemiológico y se demostró su
utilidad para el análisis genético rápido de cepas de SGB siendo capaz de detectar
heterogeneidad genómica dentro de serotipos específicos (Brandolini
et al., 2014).
Estudios
similares a nuestro trabajo fueron realizados por Brandolini et al.
(2014)
sobre casos de transmisión de SGB a través de la ingestión de leche materna.
Para su investigación utilizaron la técnica RAPD para el análisis genético de
hemocultivos, muestras de LCR y de leche materna. Los resultados revelaron
cuatro perfiles de bandas diferentes que indicaron la presencia de cuatro clones,
lo que demostró una relación entre la leche materna y la infección de los
lactantes en los cuatro casos. Además, observaron que los patrones de bandas de
SGB fueron idénticos dentro de cada conjunto de cepas, es decir, entre los
aislamientos de hemocultivo y LCR neonatal y de la leche materna, lo que indicó
la presencia del mismo clon. Por otro lado, los aislamientos obtenidos de cada
par madre-recién nacido mostraron patrones de bandas claramente diferentes,
descartando una correlación epidemiológica al igual que en el presente trabajo.
De manera similar, Nanayakkara et al., (2018), analizaron
aislamientos obtenidos de madrerecién nacido mediante la técnica RAPD, el
análisis reveló la presencia de una misma cepa de SGB en el 2,5% de los casos,
indicando una transferencia entre la madre y el recién nacido. Las
investigaciones realizadas por Nanayakkara et al., (2018) y Brandolini et
al. (2014) apoyan los resultados obtenidos en el presente trabajo donde se
detectaron cuatro patrones de bandas diferentes correspondientes a los cuatro
pares de aislamientos madre-recién nacido analizadas. Este resultado apoya la
idea que la colonización por SGB del recién nacido durante su paso por el canal
de parto es la principal causa de EOD. Por otra parte, un estudio realizado con
PFGE por Hansen et al., (2004) en aislamientos de
SGB demostró que la cepa colonizadora era homogénea y estable en cada mujer
embarazada estudiada. Los resultados indicaron que prácticamente todas las
mujeres fueron colonizadas por un solo clon de SGB y en solo unas pocas, se
encontraron dos clones diferentes simultáneamente. Para la aplicación de PFGE
utilizaron las enzimas de restricción ApaI, XhoI y NotI,
que no arrojaron resultados satisfactorios, y las enzimas SmaI o SalI,
con las que se obtuvieron patrones de bandas de buena calidad. De la misma
manera, en el presente trabajo la enzima de restricción ApaI solo logró
distinguir uno de los pares de clones, mientras que SmaI permitió
diferenciar cuatro tipos clonales correspondientes a cada par de cepas madre-recién
nacido.
Los
resultados obtenidos por Hansen et al., (2004) refuerzan la idea
de que los recién nacidos se infectan con el mismo clon que coloniza a sus madres,
a partir de la transmisión vertical de SGB. De manera similar Hirai
et al., (2020)
aplicaron la técnica PFGE para la caracterización de cepas invasivas de SGB aisladas
de recién nacidos y adultos que desarrollaron la enfermedad. Para ello, al
igual que en el presente trabajo, utilizaron la enzima de restricción SmaI.
A partir de los patrones de bandas generados identificaron clones de SGB
asociados a iguales serotipos, genotipos de resistencia y susceptibilidad a
antibióticos, concluyendo la existencia de una transmisión horizontal en la
comunidad durante 10 años.
El
presente trabajo concuerda con otros investigadores en que RAPD es una técnica
rápida, fácil y económica de realizar en comparación con los procedimientos
alternativos para el análisis de ADN. Además, consideramos que la técnica de
RAPD es suficientemente robusta para ser aplicada como una técnica analítica de
ADN para investigaciones epidemiológicas de rutina (Martinez
et al., 2000;
Brandolini
et al., 2014;
Nanayakkara
et al., 2018).
Si bien, Martinez et al. (2000) sugieren que la tipificación RAPD generada
por la combinación de cebadores OPS11, OPB17 y OPB18 aumenta la capacidad de la
metodología para detectar la variabilidad entre aislamientos, a partir de los
valores obtenidos para D y los patrones de bandas observados en el presente
trabajo, sostenemos que la aplicación individual del cebador OPS11 es capaz de diferenciar
los pares de cepas madre-recién nacido. Como se expuso anteriormente, la
técnica de PFGE continúa siendo la técnica gold estándar elegida para estudios
epidemiológicos, por lo que también fue incluida en nuestro análisis (Åberg
et al., 2019).
En este sentido, la obtención de resultados similares por medio de las técnicas
RAPD y PFGE aplicadas en el presente trabajo, nos permite proponer a la técnica
RAPD como una alternativa para su aplicación en laboratorios de baja y mediana
complejidad. Finalmente, cabe destacar la importancia y trascendencia desde un
enfoque clínico-epidemiológico de estos aislamientos parentales debido a la
escasez de situaciones diagnósticas que permiten recuperar SGB en simultáneo
tanto de la madre como de su recién nacido.
Esto
se debe fundamentalmente a que a partir de la implementación de la búsqueda
obligatoria de SGB por Ley Nacional N° 26.369 en toda mujer embarazada entre 35-37
semanas de gestación, si se detecta colonización materna se implementa la PIP y
se previene la infección neonatal. Por lo tanto, los cuatro aislamientos parentales
recuperados en este estudio son valiosos y representativos para evaluar la
transmisión vertical. Este es el primer estudio en la región y en el país que ha
optimizado la técnica RAPD para su aplicación en el estudio de SGB dentro de
centros de salud y universidades de Argentina y países vecinos como Paraguay y
Brasil. A futuro se espera que esta técnica sea reproducida en otros
laboratorios para la obtención de un mayor número de datos epidemiológicos que
permitan estudios más complejos sobre dispersión clonal de cepas de SGB en embarazadas
e infecciones invasivas en recién nacidos.
CONCLUSIONES
Se
seleccionó el cebador OPS11 por su robustez y capacidad para discriminar cepas
de SGB no relacionadas a partir de la técnica RAPD. Se confirmó la transmisión
vertical y la relación clonal de SGB de las madres estudiadas a sus respectivos
recién nacidos, por técnica de RAPD y por PFGE, que es la técnica de
referencia, encontrando perfiles de bandas idénticos para cada par de
aislamientos madre-recién nacido. Se encontraron condiciones apropiadas para la
técnica RAPD, que resulta de utilidad para realizar estudios epidemiológicos
ligados a la portación materna de SGB y su transmisión al recién nacido, así
como a la eventual transmisión horizontal de esta bacteria en centros de salud.
ASPECTOS
ÉTICOS-REGULATORIOS
Este
trabajo fue aprobado por el Comité Científico del Hospital Central “Dr. Ramón
Madariaga” de la ciudad de Posadas, Misiones y por el Comité Institucional de
Ética de la Investigación en Salud de los Hospitales Materno Provincial, Materno
Neonatal y Misericordia de la ciudad de Córdoba, Córdoba. Se obtuvo
consentimiento informado por escrito para cada paciente y datos médicos
confidenciales según protocolo de estudio: C10 “Prevalencia de colonización vaginal
y rectal de Streptococcus betahemolítico grupo B (SGB o Streptococcus
agalactiae) en mujeres embarazadas de 35-37 semanas de gestación”. Este
estudio sólo revisó a pacientes humanos, ningún animal participó en ningún
aspecto del estudio. Los autores afirman que conocen las normas bioéticas vigentes
en la Argentina y en la Declaración de Helsinki y sus enmiendas.
AGRADECIMIENTOS
A
los integrantes de la Cátedra de Bacteriología de la Facultad de Ciencias
Exactas, Químicas y Naturales de la Universidad Nacional de Misiones,
especialmente a su ex Profesora Titular la Mgter. Marta Vergara por ser pionera
en este tema en la provincia de Misiones. A la Mgter. Viviana Villalba, Jefa
del Servicio de Bacteriología del Laboratorio del Hospital Escuela de Agudos
“Dr. Ramón Madariaga” por su colaboración en el aislamiento e identificación de
las cepas de Streptococcus agalactiae recuperadas de neonatos. A la Dra.
Adriana Limansky, Profesora Asociada de Bacteriología del Instituto de Biología
Molecular y Celular de Rosario, Departamento de Microbiología, Facultad de
Ciencias Bioquímicas y Farmacéuticas, Universidad Nacional de Rosario, por su
asesoramiento en la puesta a punto y ejecución de la técnica de RAPD.
REFERENCIAS
Åberg E., Ottosson A., Granlund M.,
Saeedi B., Stamm C., Brune T., Tammelin A., Johansson S. (2019) Harbouring
group B streptococci in a neonatal intensive care unit led to an outbreak among
preterm infants. Acta Paediatr. 108(1): 58-61.
Barcaite E., Bartusevicius A., Tameliene
R., Kliucinskas M., Maleckiene L., Nadisauskiene R. (2014) Prevalence of
maternal group B streptococcal colonisation in European countries. Acta Obstet.
Gynecol. Scand. 87: 260-271.
Brandolini M., Corbella M., Cambieri
P., Barbarini D., Sassera D., Stronati M., Marone P. (2014) Late-onset neonatal
group B streptococcal disease associated with breast milk transmission:
molecular typing using RAPD-PCR. Early Hum. Dev. 90: S84-S86.
Cariaga Martinez A.E., Zapata P.D.
(2007) Protocolos
de Extracción de ADN. En: El Laboratorio de Biología Molecular Universitaria de
Misiones. Editorial Universitaria, Misiones, Argentina, pp. 23-39.
Centers for Disease Control and
Prevention (CDC) (1996) Guía del CDC para quimioprofilaxis intraparto. MMWR
Recomm. Rep. 45: 16-17.
Centers for Disease Control and Prevention
(CDC) (2002) Prevention of perinatal group B streptococcal disease. MMWR Recomm. Rep .51:
1-22.
Centers for Disease Control and
Prevention (CDC) (2010) Prevention of perinatal group B: revised guidelines
from CDC. MMWR Recomm. Rep. 59 (RR-10):1-31.
Chatellier S., Huet H., Kenzi S.,
Rosenau A., Geslin P., Quentin R. (1996) Genetic diversity of rRNA operons of
unrelated Streptococcus agalactiae strains isolated from cerebrospinal
fluid of newborns suffering from meningitis. J. Clin. Microbiol. 34:2741-7.
Chen V.L., Avci F.Y., Kasper D.L.
(2013) A maternal vaccine against group B Streptococcus: past, present, and
future. Vaccine. 31: 13-19.
Collin S.M., Lamb P., Jauneikaite
E., Le Doare K., Creti R., Berardi A., Heath P.T., Sriskandan S., Lamagni T.
(2019) Hospital clusters of invasive Group B Streptococcal disease: A systematic
review. J. Infect. 79(6): 521-527.
Do Nascimento C.S., dos Santos N.F.,
Ferreir R.C., Taddei C.R. (2019) Streptococcus agalactiae in pregnant
women in Brazil: prevalence, serotypes, and antibiotic resistance. Braz. J. Microbiol.
50(4): 943-952.
Faccone D., Lalonardi F., Abel S.,
Machain M., Errecalde L., Littvik A., Kauffman S., Galas M., WHONET-Argentina
Group; Corso A. (2010) Multiple-Clones of Streptococcus agalactiae harbouring
lnuB gene. J Infect Dev Ctries. 4(9):580-2.
Hanna M., Noor A. Streptococcus
Group B. https://www.ncbi.nlm.nih.gov/books/NBK553143/ (Acceso: mayo de
2020).
Hansen S.M., Uldbjerg N., Kilian M.,
Sørensen U.B. (2004) Dynamics of Streptococcus agalactiae colonization
in women during and after pregnancy and in their infants. J. Clin. Microbiol. 42(1): 83-9.
Herbert M.A., Beveridge C.J., Saunders
N.J. (2004) Bacterial virulence factors in neonatal sepsis: group B Streptococcus.
Curr. Opin. Infect. Dis. 17: 225-9.
Hirai N., Kasahara K., Nakano R.,
Ogawa Y., Suzuki Y., Ogawa M., Hishiya N., Nakano A., Ichimura S., Yano H., Yoshikawa
M. (2020) Clinical characteristics and molecular epidemiology of invasive Streptococcus
agalactiae infections between 2007 and 2016 in Nara, Japan. Plos one. 15(10),
e0240590.
Huet H., Martin C., Geslin P., Grimont
F., Quentin R. (1993) Ribotyping of Streptococcus agalactiae strains
isolated from vaginas of asymptomatic women. Res. Microbiol. 144: 457-65.
Hunter P.R., Gaston M.A. (1998) Numerical
index of the discriminatory ability of typing systems: an application of
Simpson’s index of diversity. J. Clin.
Microbiol. 26(11): 2465-6.
Idil N., Bilkay I.S. (2014) Application
of RAPD-PCR for determining the clonality of methicillin resistant Staphylococcus
aureus isolated from different hospitals. Braz Arch. Biol. Technol. 57(4):
548-553.
Ley Nacional N° 26.369. (2008) Examen obligatorio
de detección del estreptococo Grupo B agalactiae a todas las embarazadas. Boletín Oficial N° 31399. Argentina.
Manning S.D. (2003) Molecular
epidemiology of Streptococcus agalactiae (group B Streptococcus).
Front. Biosci. 1:1-18.
Manrique Martín G., García-Martín
A., Rincón-López E., Saavedra-Lozano J. (2020) Streptococcus agalactiae: causa
de artritis séptica inusual a partir de los 3 meses de edad. Caso clínico. Arch. Argent. Pediatr. 118(4): e392-e395.
Martinez G., Harel J., Higgins R.,
Lacouture S., Daignault D., Gottschalk M. (2000) Characterization of Streptococcus
agalactiae isolates of bovine and human origin by randomly amplified
polymorphic DNA analysis. J. Clin. Microbiol. 38(1):
71-8.
Nanayakkara D., Liyanapathirana V.,
Kandauda C., Gihan C, Ekanayake A., Adasooriya D. (2018) Maternal vaginal
colonization with selected potential pathogens of neonatal sepsis in the era of
antimicrobial resistance, a single center experience from Sri Lanka. BMC
Infect. Dis. 18(1): 351.
Patras K.A., Rösler B., Thoman M.L.,
Doran K.S. (2015) Characterization of host immunity during persistent vaginal
colonization by Group B Streptococcus. Mucosal Immunol. 8(6): 1339-48.
Quentin R., Huet H., Wang F.S., Geslin
P., Goudeau A, Selander R.K. (1995) Characterization of Streptococcus
agalactiae strains by multilocus enzyme genotype and serotype:
identification of multiple virulent clone families that cause invasive neonatal
disease. J. Clin. Microbiol. 33(10):2576-81.
Raabe V.N., Shane A.L. (2019) Group
B Streptococcus (Streptococcus agalactiae). Microbiol. Spectr. 7:
10.1128.
Rolim L.D.N., Cavalcante M.A.D.Q.,
Urben A.F., Buso G.S.C. (2011) Use of RAPD molecular markers on differentiation
of Brazilian and Chinese Ganoderma lucidum strains. Braz. Arch. Biol.
Technol. 54(2): 273-281.
Russell N.J., Seale A.C., O’Driscoll
M., O’Sullivan C., et al. (2017) Maternal colonization with group B Streptococcus
and serotype distribution worldwide: systematic review and meta-analyses. Clin.
Infect. Dis. 65(S2): S100-111.
Sarrión-Sos N., Morell-García M.,
Martínez- Sebastián L., Centeno-Rubiano J.M., Montesinos-Sanchis E., Orta-Sibú N.
(2018) Síndrome
celulitis-adenitis, una forma infrecuente de presentación de la sepsis neonatal
tardía: A propósito de dos casos. Arch Argent
Pediatr. 769-772.
Shadbad M.A., Kafil H.S., Rezaee M.A.,
Farzami M.R., Dehkharghani A.D., Sadeghi J., Gholizadeh P., Khodaei F., Aghazadeh
M. (2020) Streptococcus agalactiae clinical isolates in Northwest Iran:
antibiotic susceptibility, molecular typing, and biofilm formation. GMS Hyg. Infect.
Control. 15.
Szymusik I., Kosińska-Kaczyińska
K., Pietrzak B., Wielgoś M. (2014) Do we need a different approach to GBS
screening? Ginekol. Pol. 85: 456-60.
Taniyama D., Maruki T., Maeda T.,
Yoshida H., Takahashi T. (2020) Repetitive cellulitis caused by Streptococcus
agalactiae isolates with different genotypic and phenotypic features in a
patient having upper extremity with lymphedema after mastectomy and axillary lymph
node dissection. IDCases. e00793.
Toresani I., Limansky A., Bogado I.,
Guardati M.C., Viale A., Sutich E.G. (2001) Phenotypic and genotypic study of Streptococcus
agalactiae in vagina of pregnant women in Argentina. Medicina-B Aires. 61(3):295-300.



